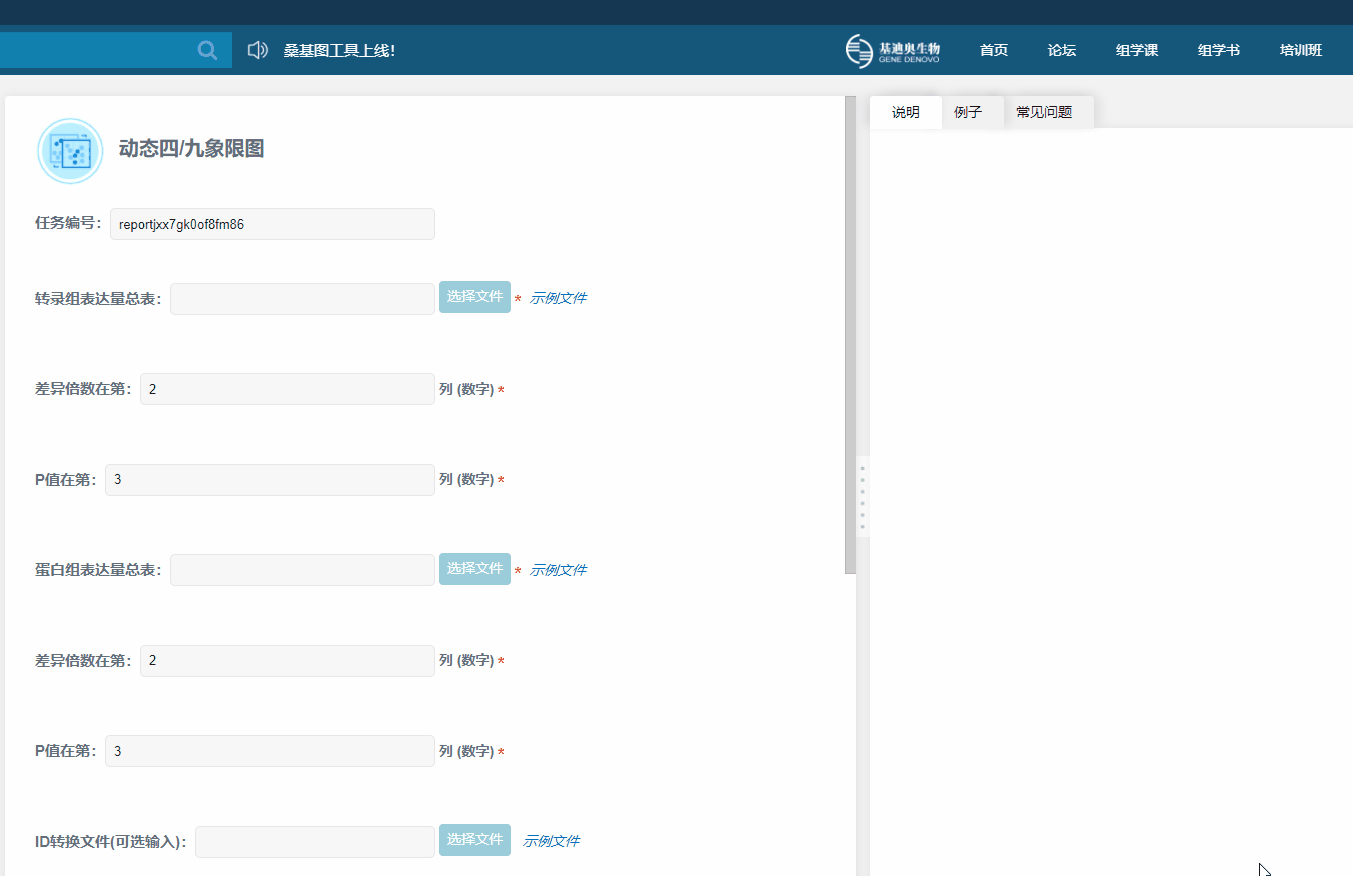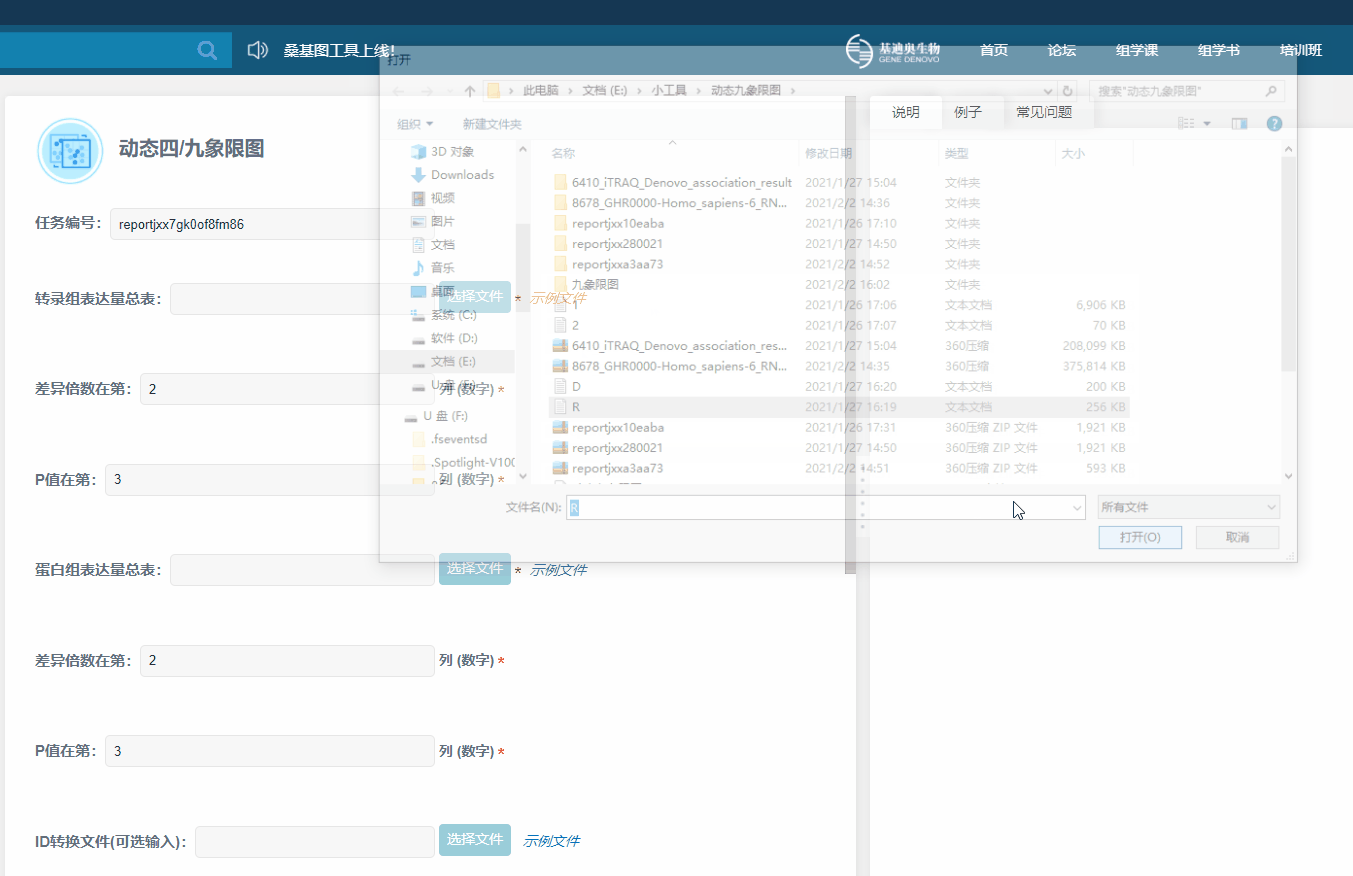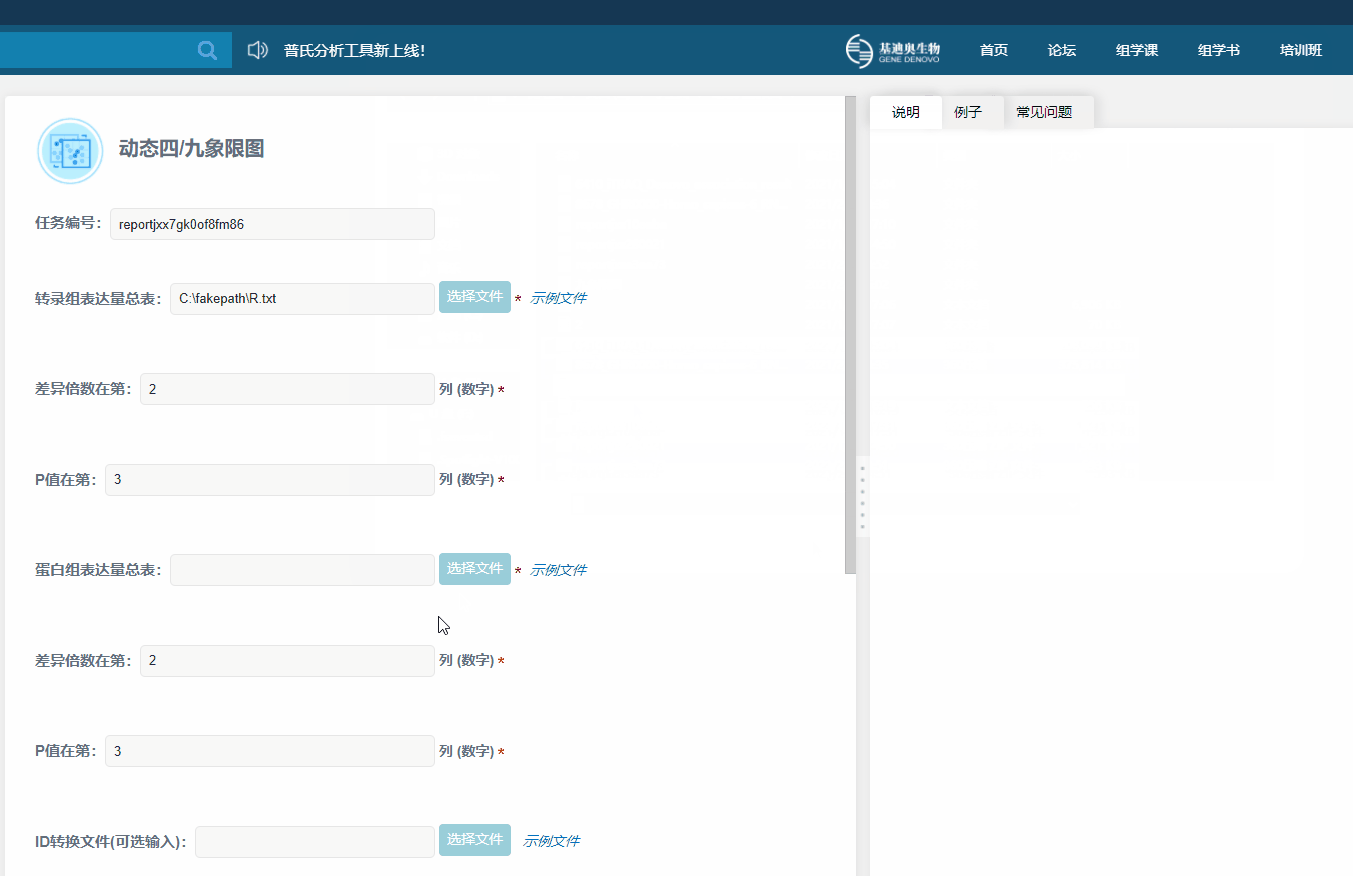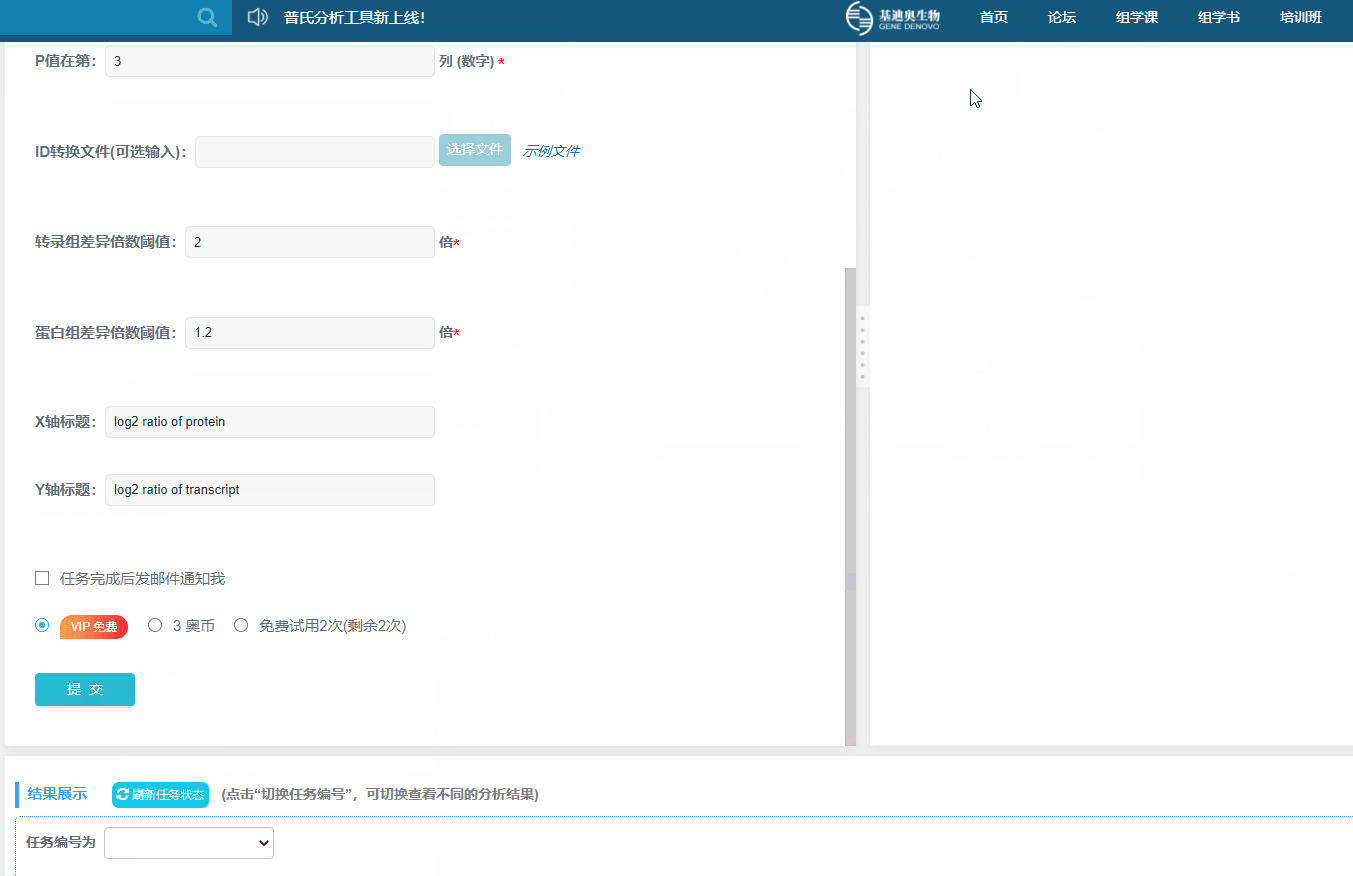
新建任务
任务切换
全局修改
参数修改
差异分析
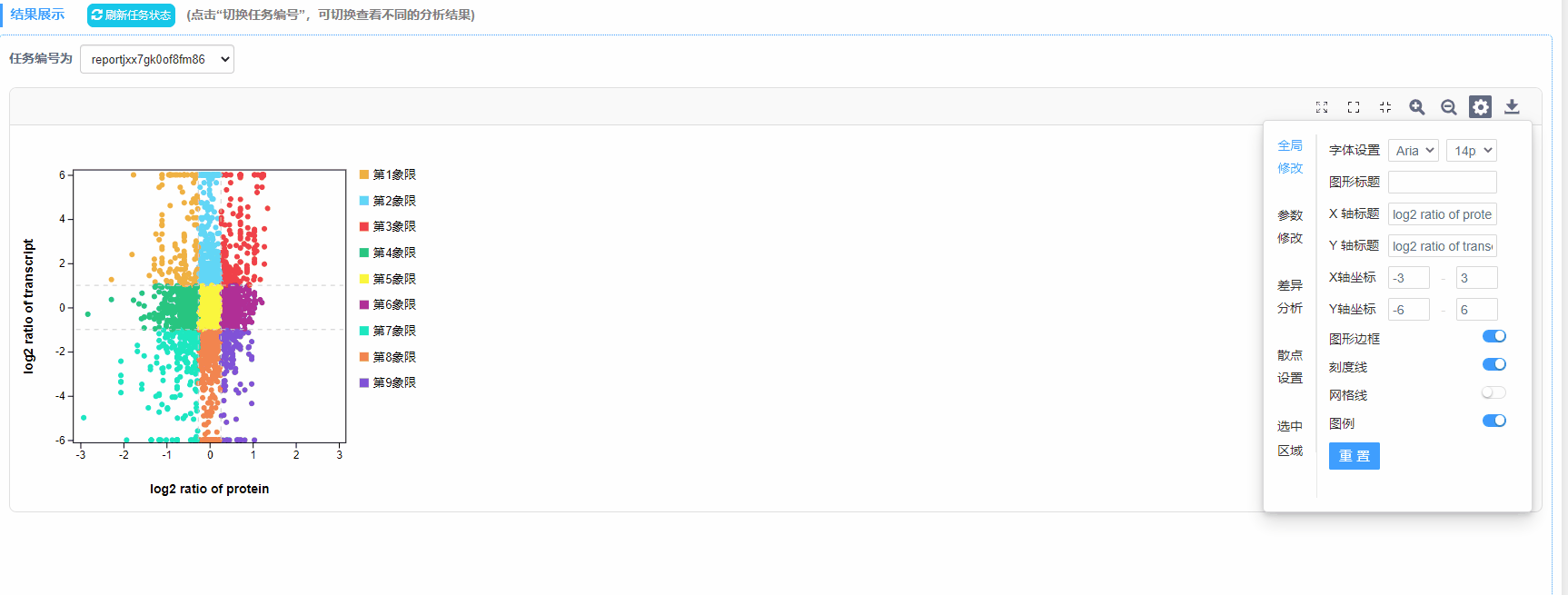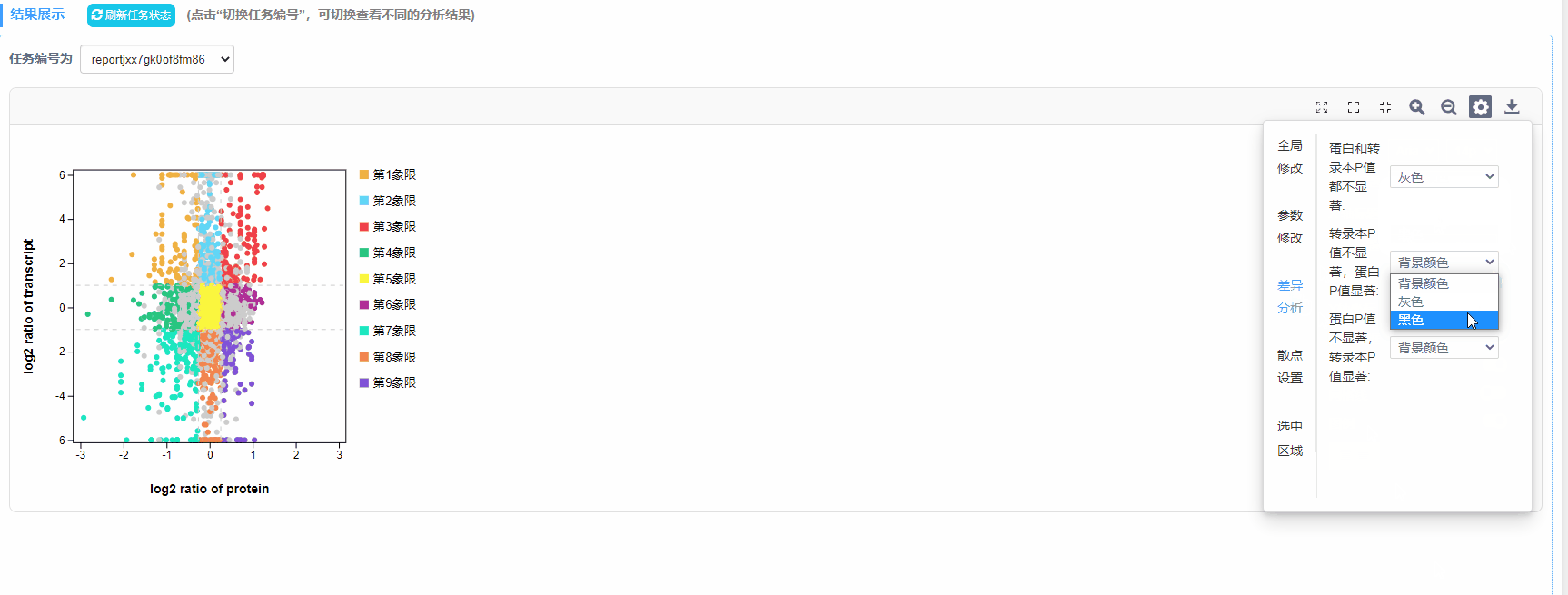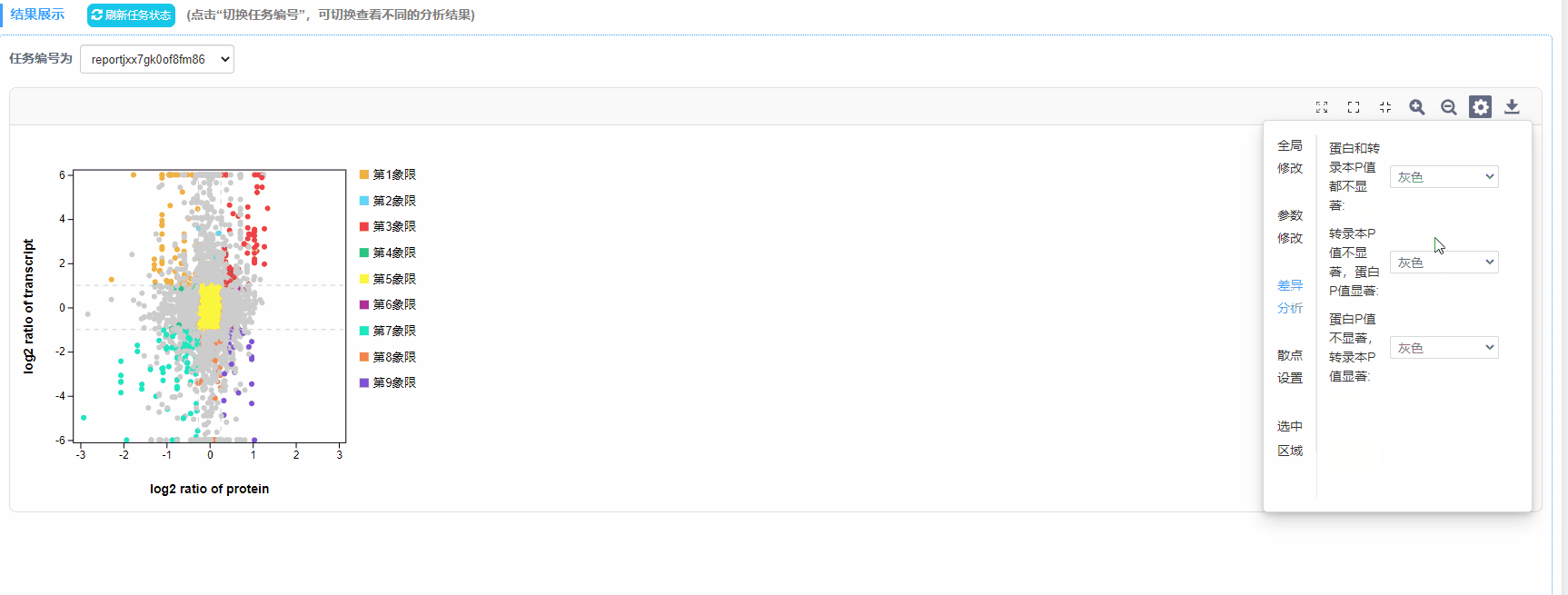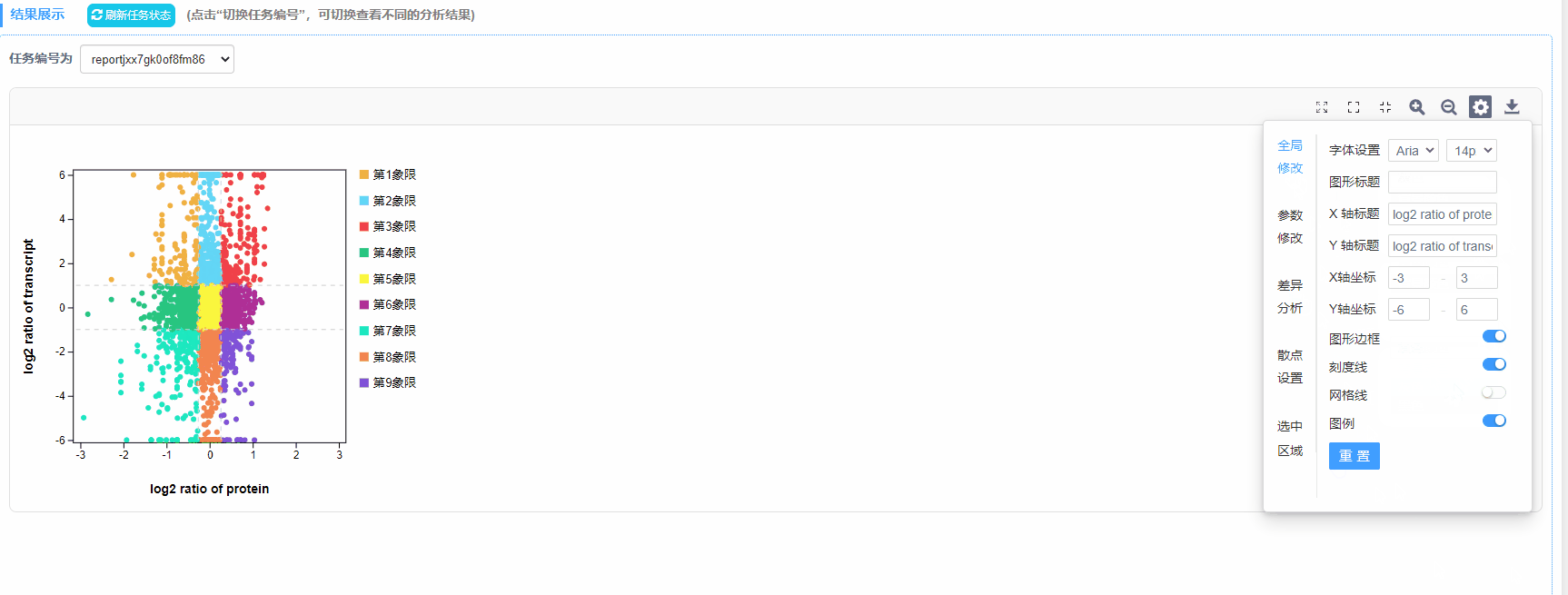
散点设置
功能:
用于转录组与蛋白组关联分析。
输入:
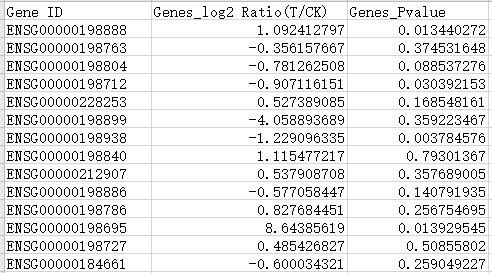
1)必要文件1:转录组表达量总表,必须包含基因ID,组间表达量差异倍数;必须需将差异倍数在表格的第几列标明出来,如在表格第二列,则输入2。如需要筛选差异显著性基因,输入表格中必须包含显著性P值,并标注在表格第几列。
示例文件:转录组表达量总表,差异倍数在表格第2列,差异显著性P值在第3列。
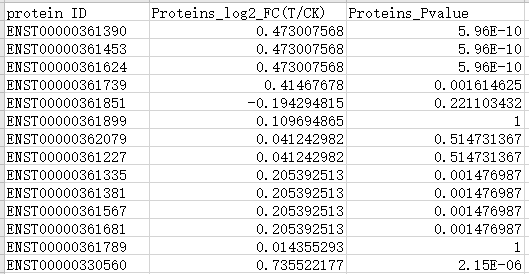
2)必要文件2:蛋白组表达量总表,必须包含蛋白ID和组间表达量差异倍数;需将差异倍数在表格的第几列标明出来,如在表格第二列,则输入2。如需要筛选差异显著性蛋白,输入表格中必须包含显著性P值,并标注在表格第几列。
示例文件:蛋白组表达量总表,差异倍数在表格第2列,差异显著性P值在第3列。
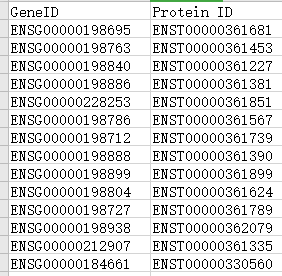
3)选择输入文件:如基因和蛋白的ID不一样,可添加ID对应文件,用于将基因和蛋白的ID对应。
参数:
1)转录组差异倍数阈值:自定义(默认2倍)
2)蛋白组差异倍数阈值:自定义(默认1.2倍)
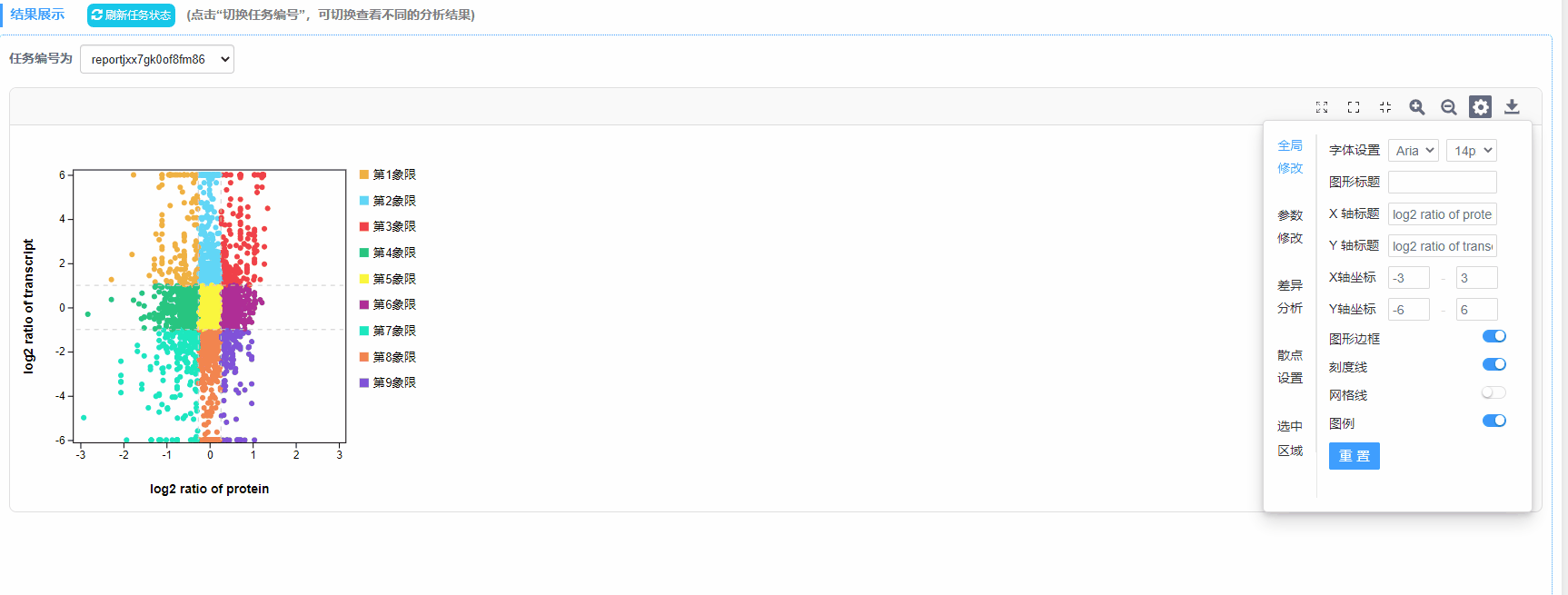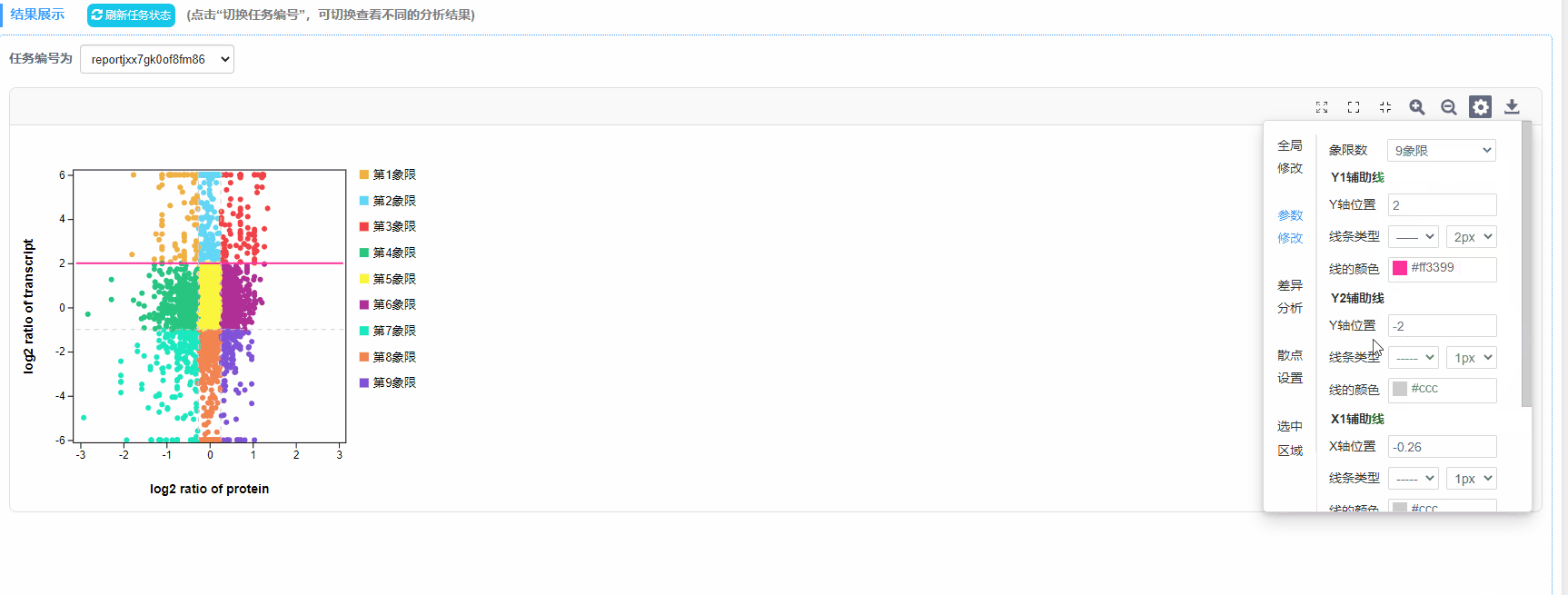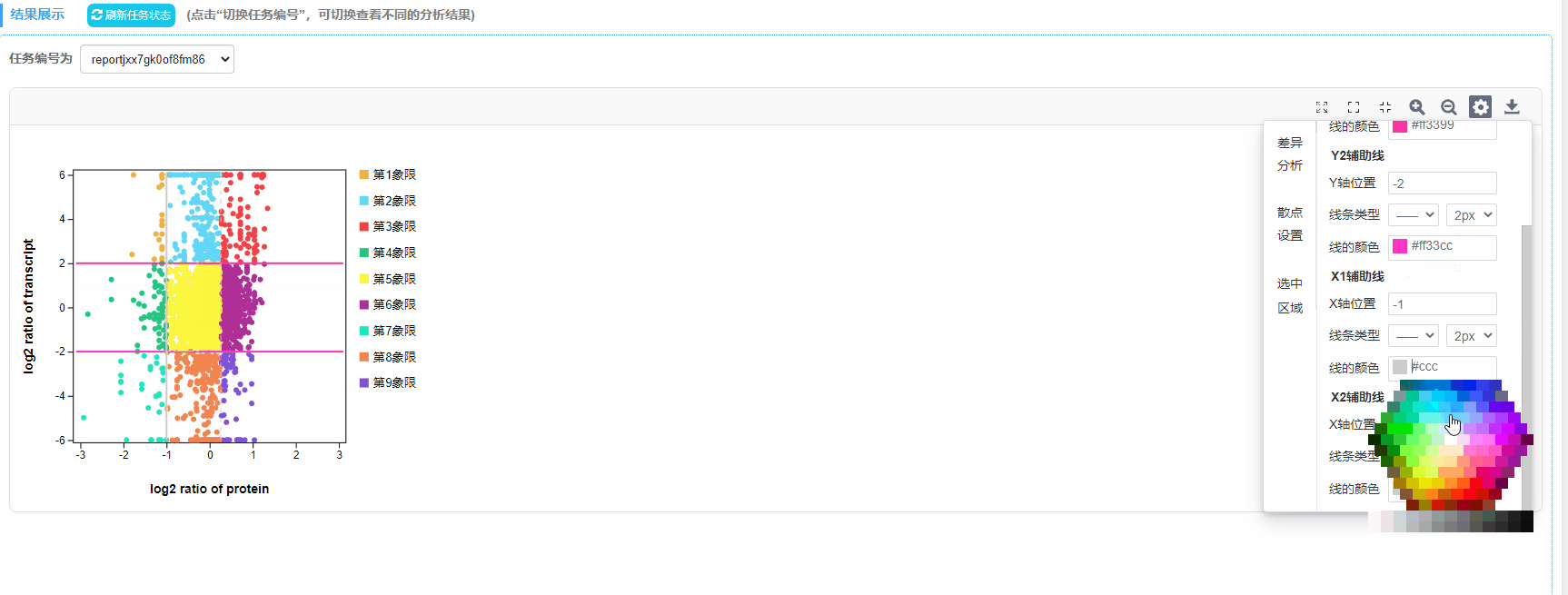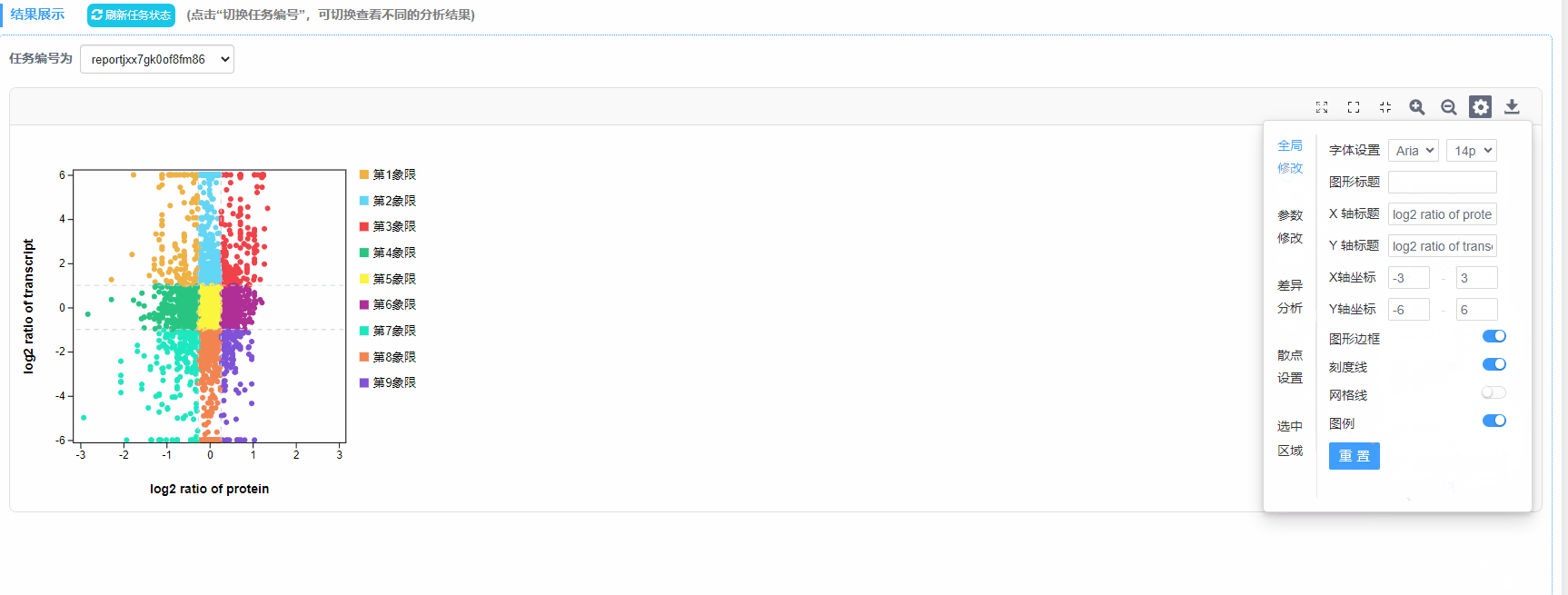
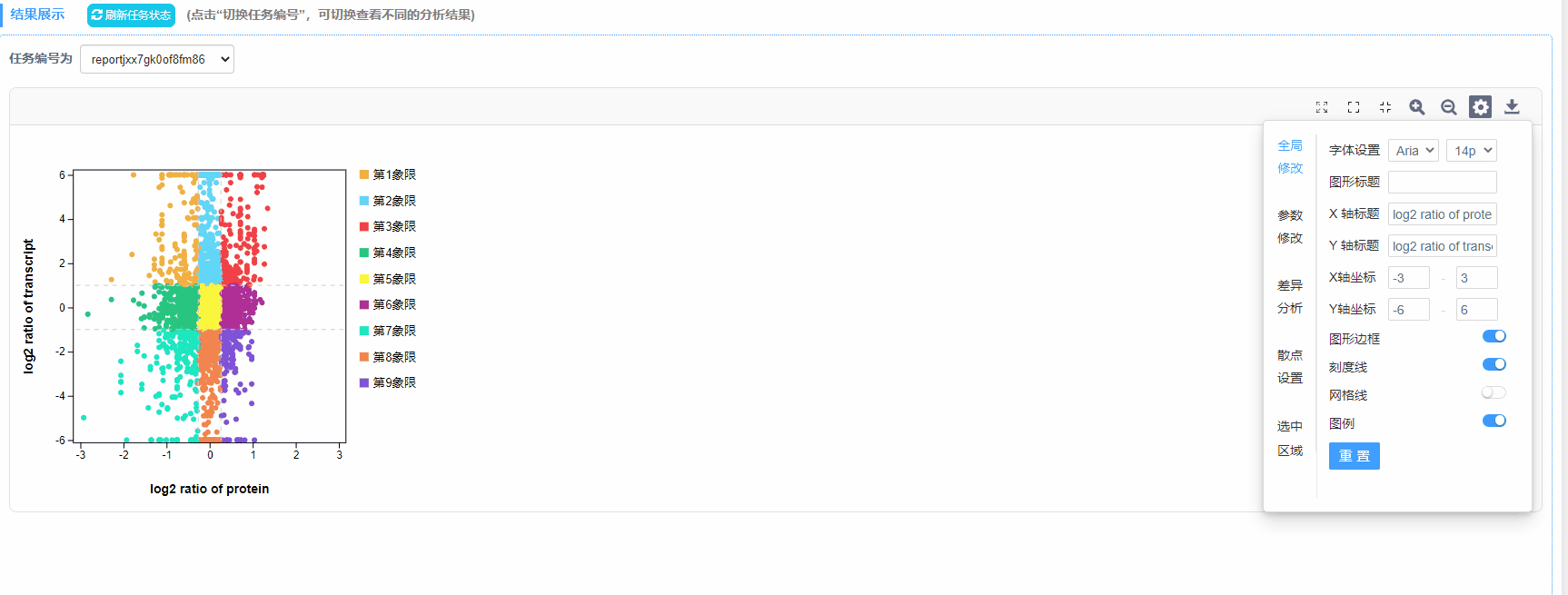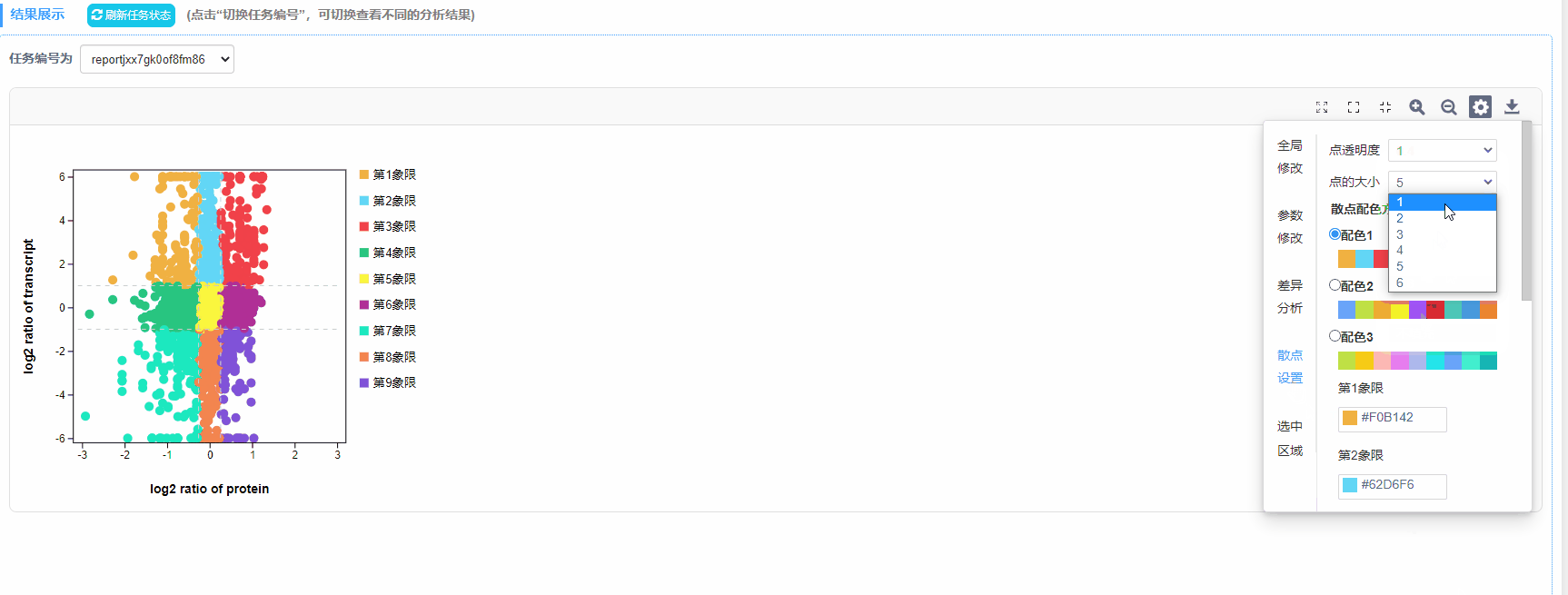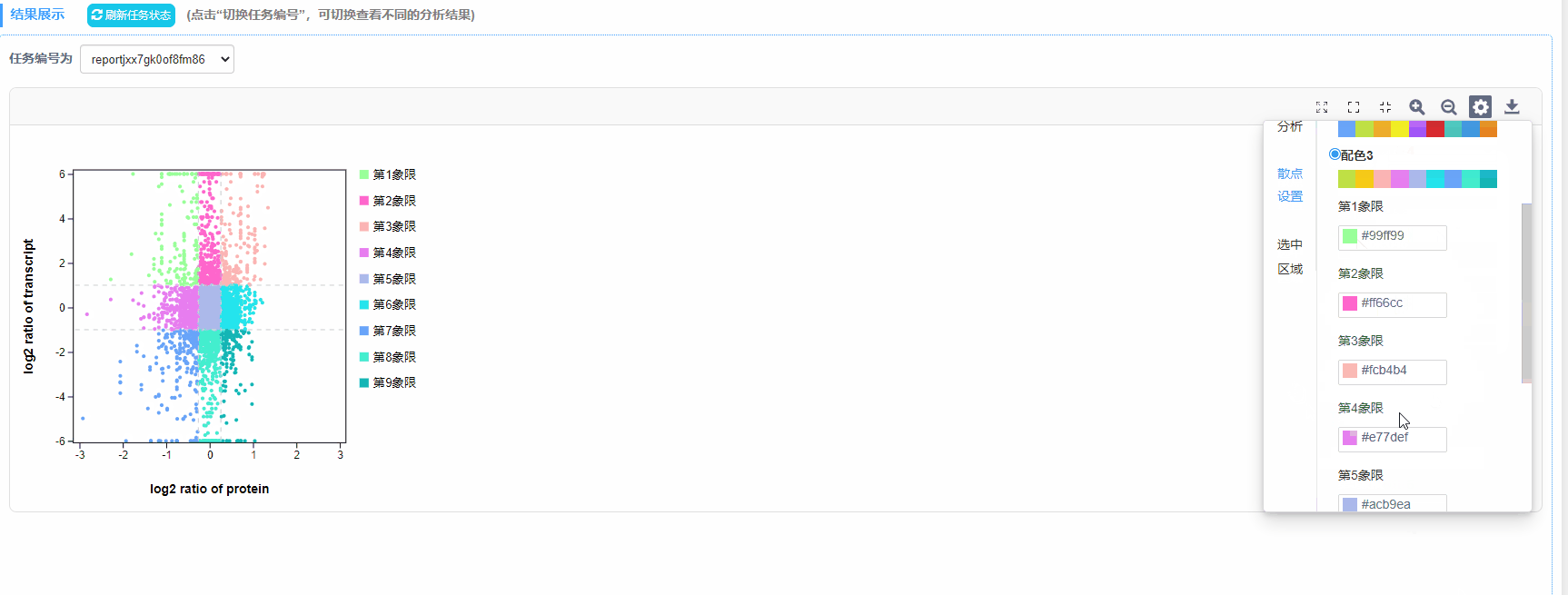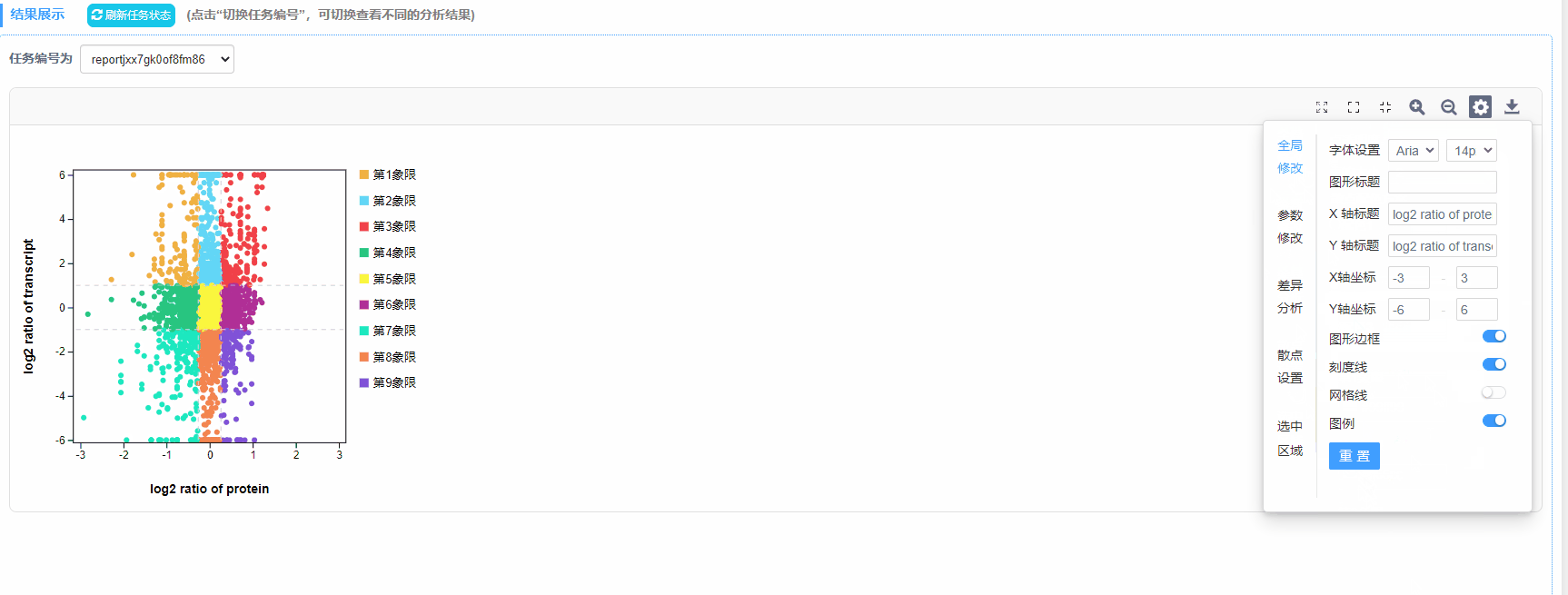
3)X轴标题:自定义(默认为log2 ratio of protein)
4)Y轴标题:自定义(默认为log2 ratio of transcript)
动态参数:
1)图形标题:自定义
2)X/Y坐标轴:自定义范围
3)图形边框/刻度线/网格线/图例:设置是否展示
4)象限数:四/九象限
5)X/Y辅助线:设置辅助线坐标位置,线条类型,粗细以及线的颜色
6)差异分析:筛选差异倍数阈值范围内P值不显著的散点
7)散点:透明度选择、颜色修改
8)选中区域:点/名称大小和颜色设置
输出:
1)数据1:四、九象限总表,包含所有基因/蛋白所在的象限信息,以及表达量信息。
2)数据2:各象限分表(分别为4个和9个),包含各自象限的基因/蛋白表达量信息。
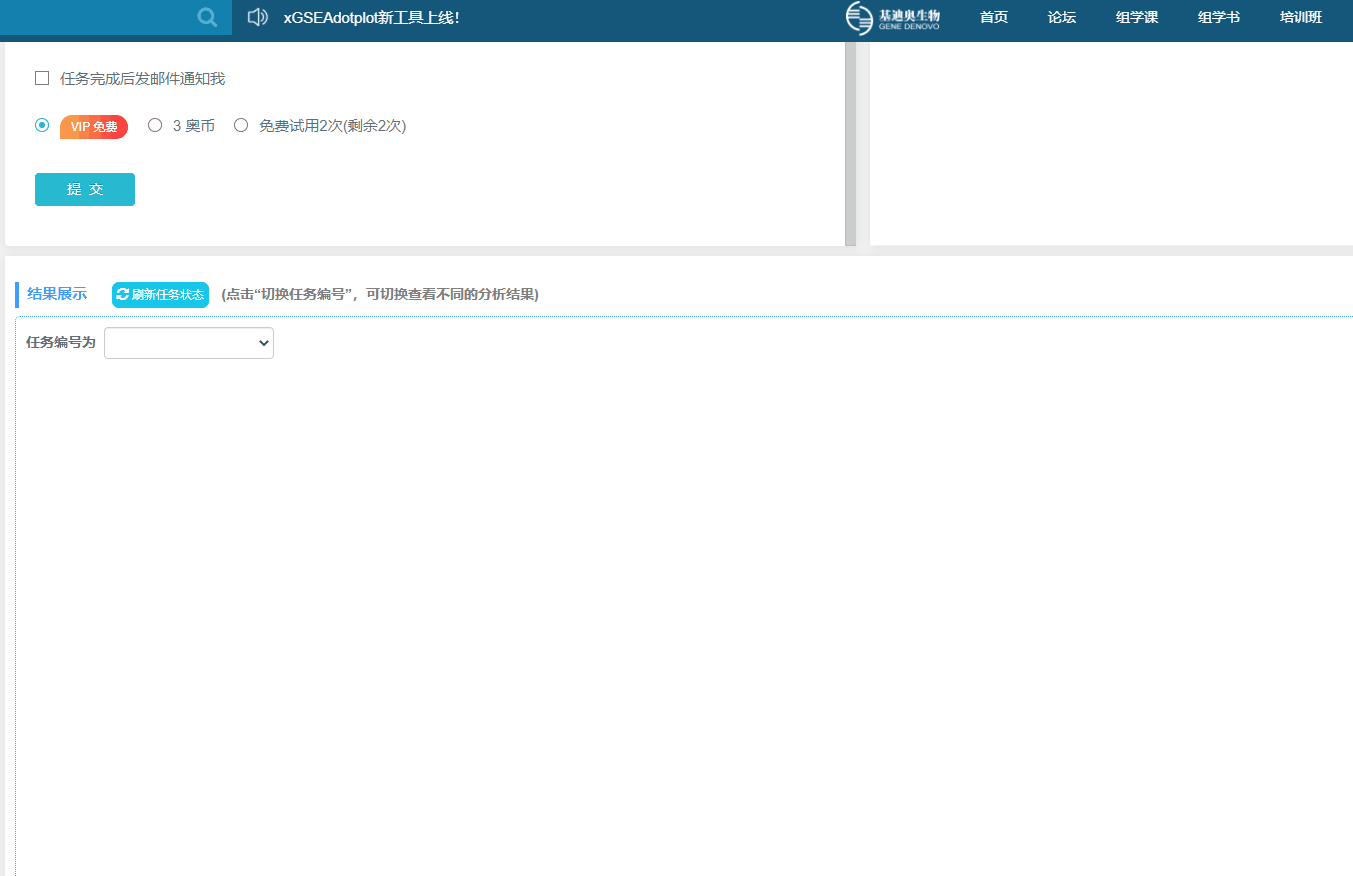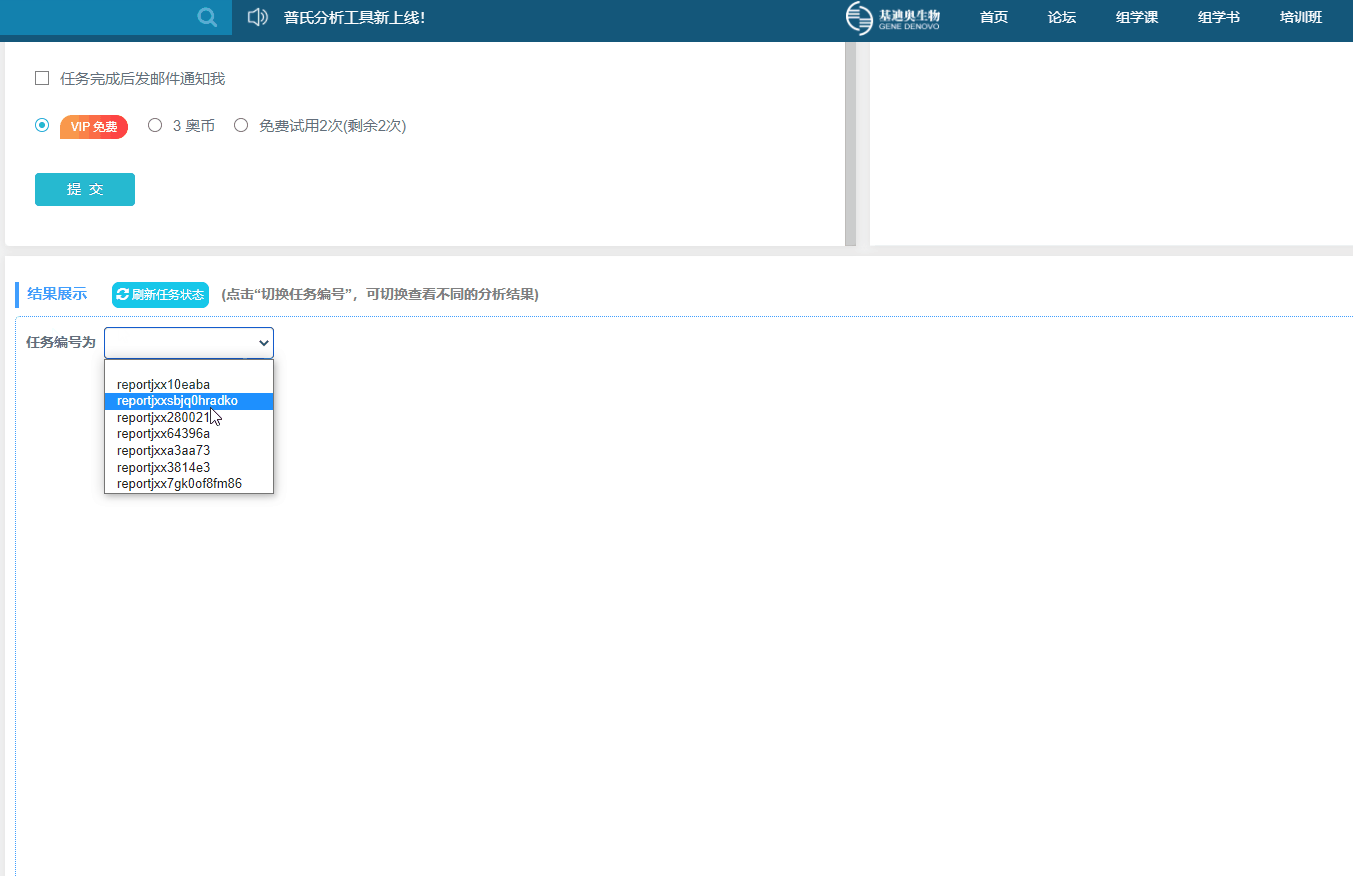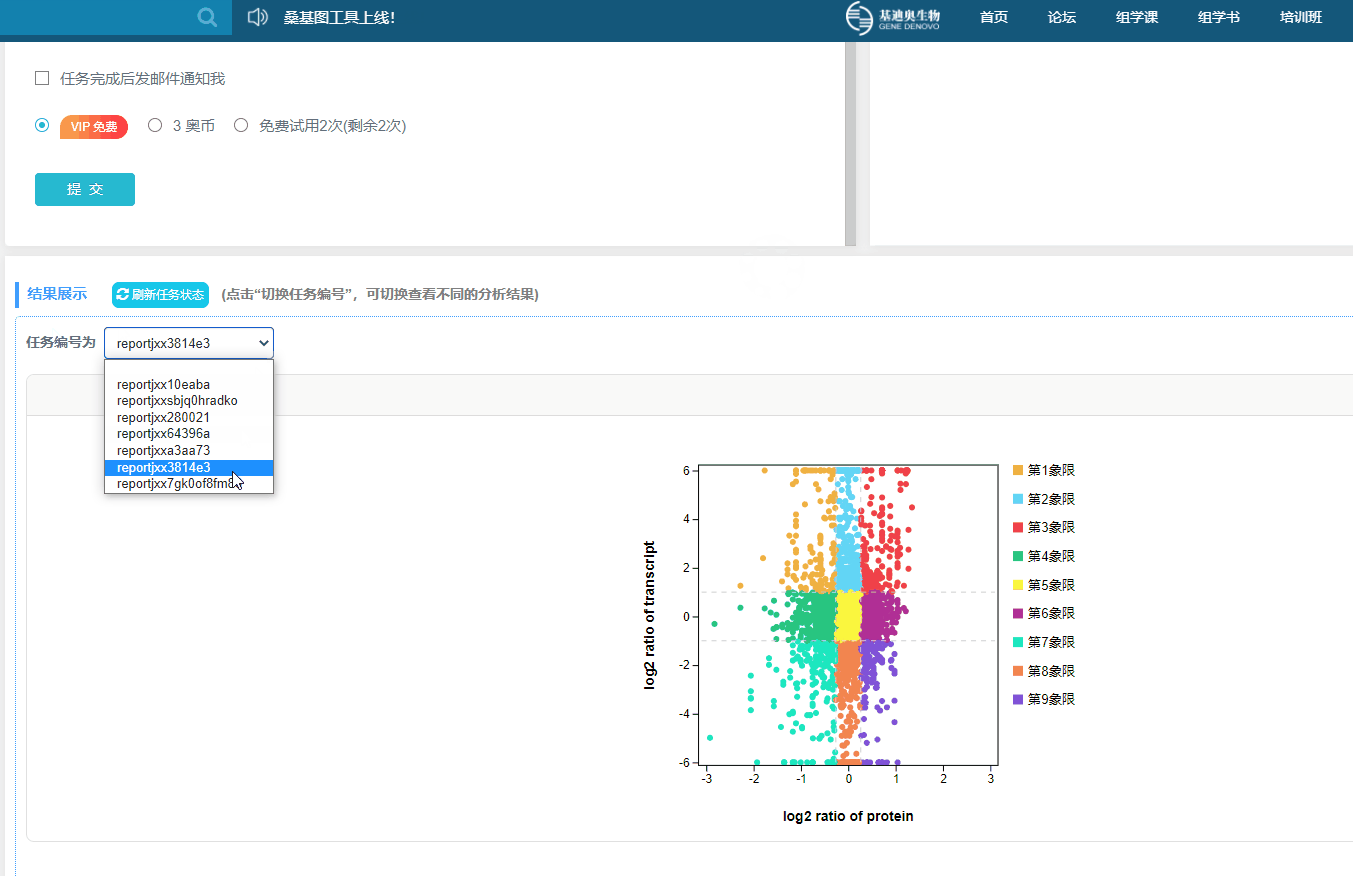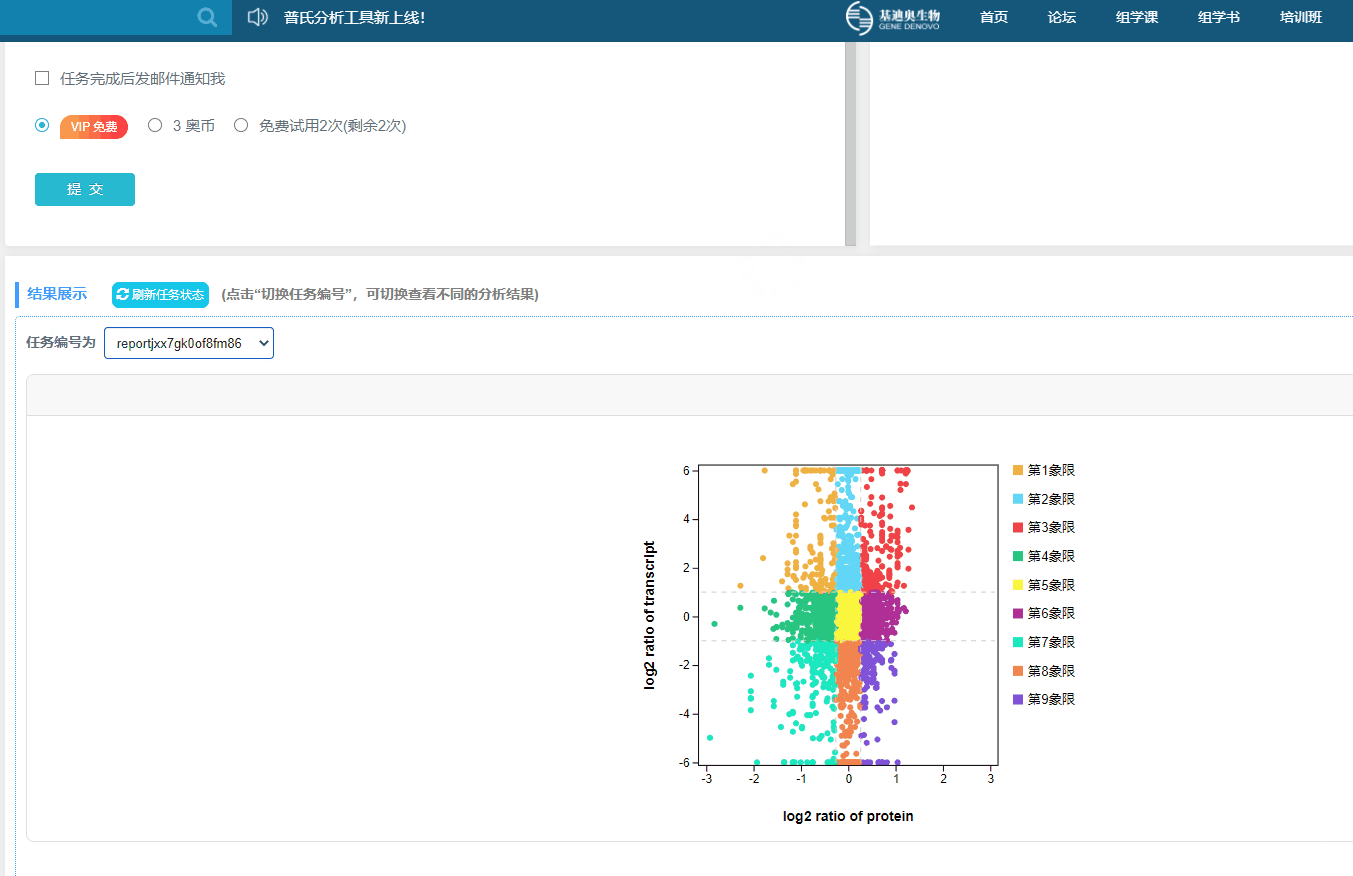
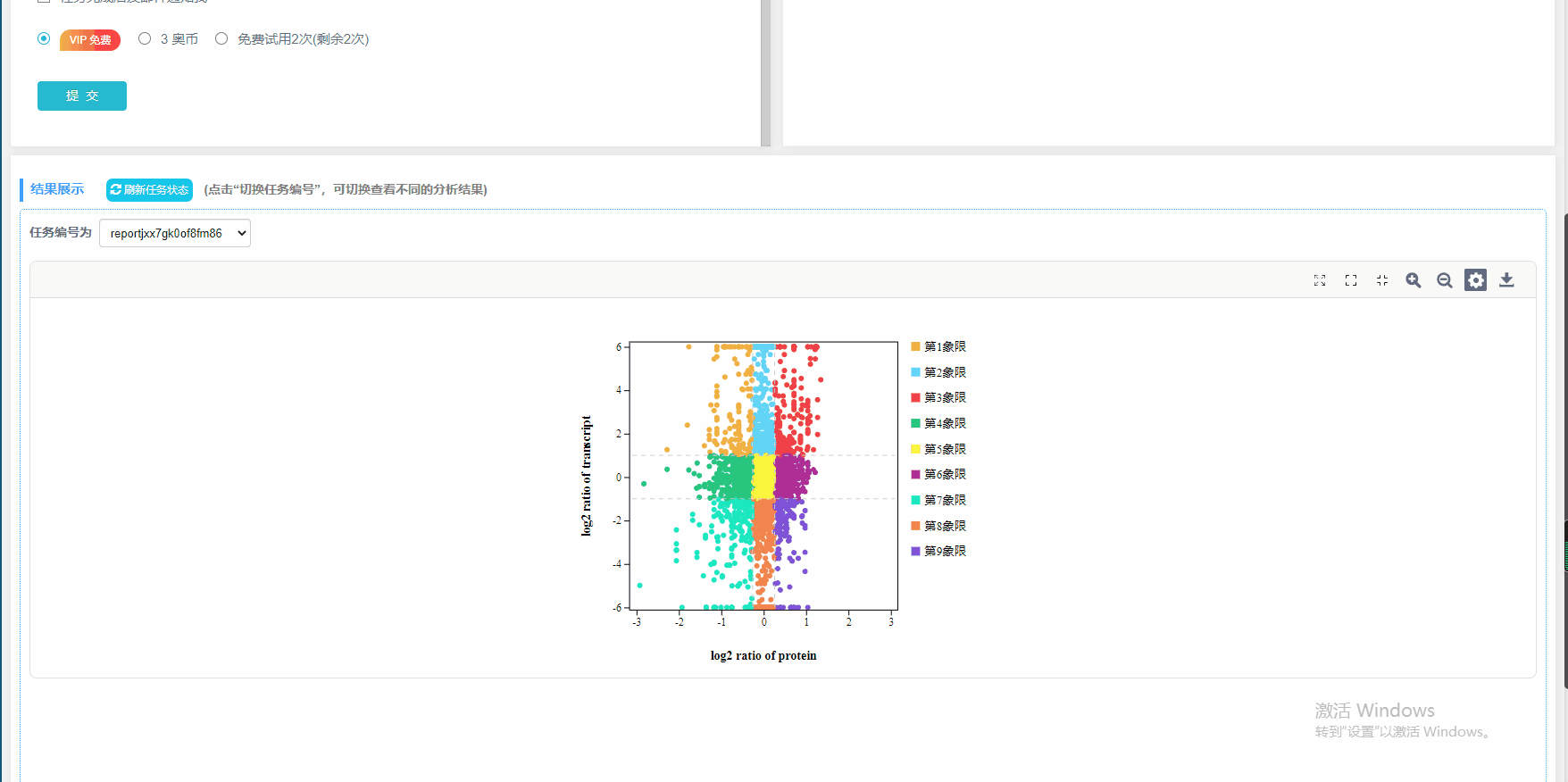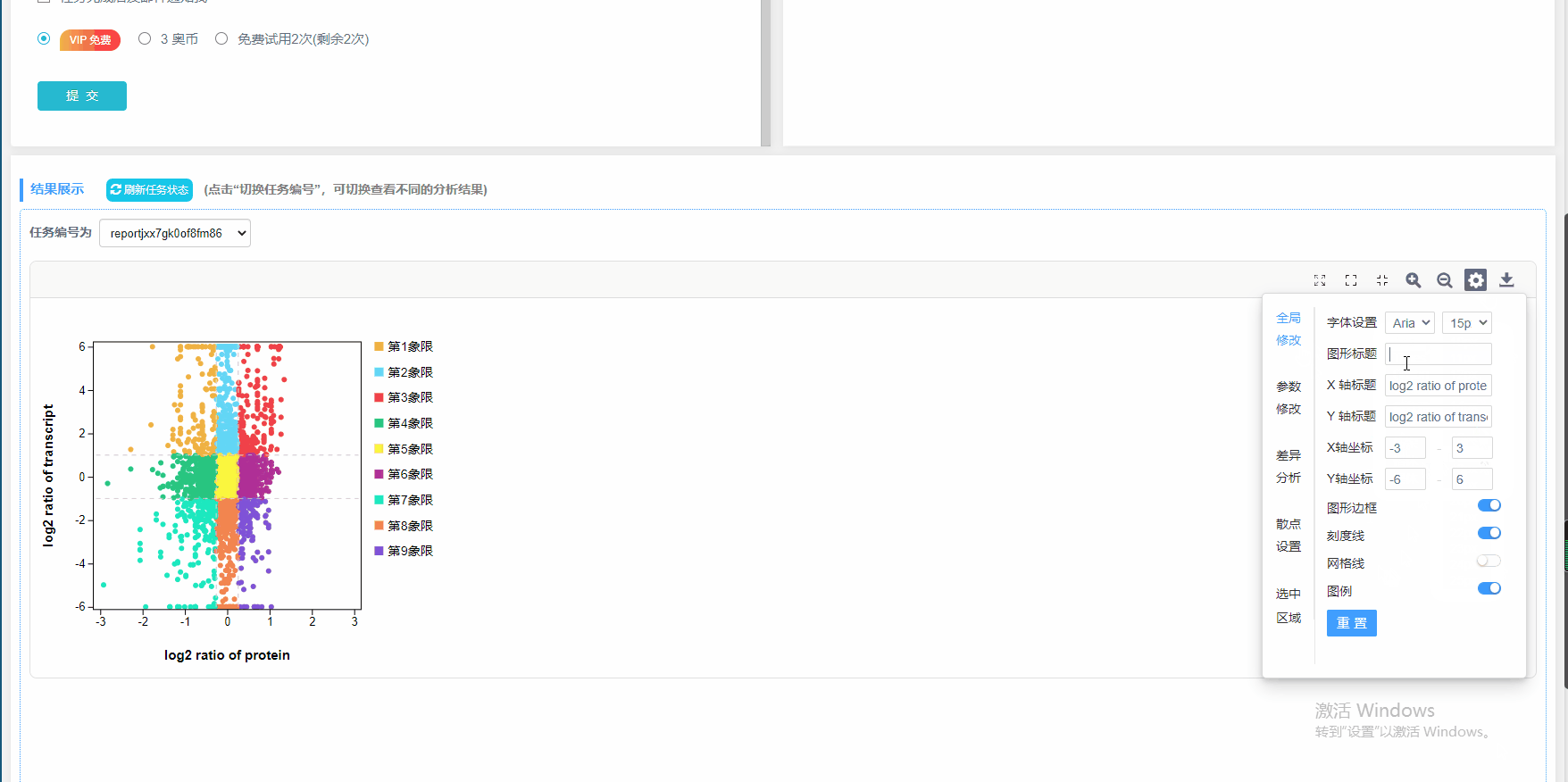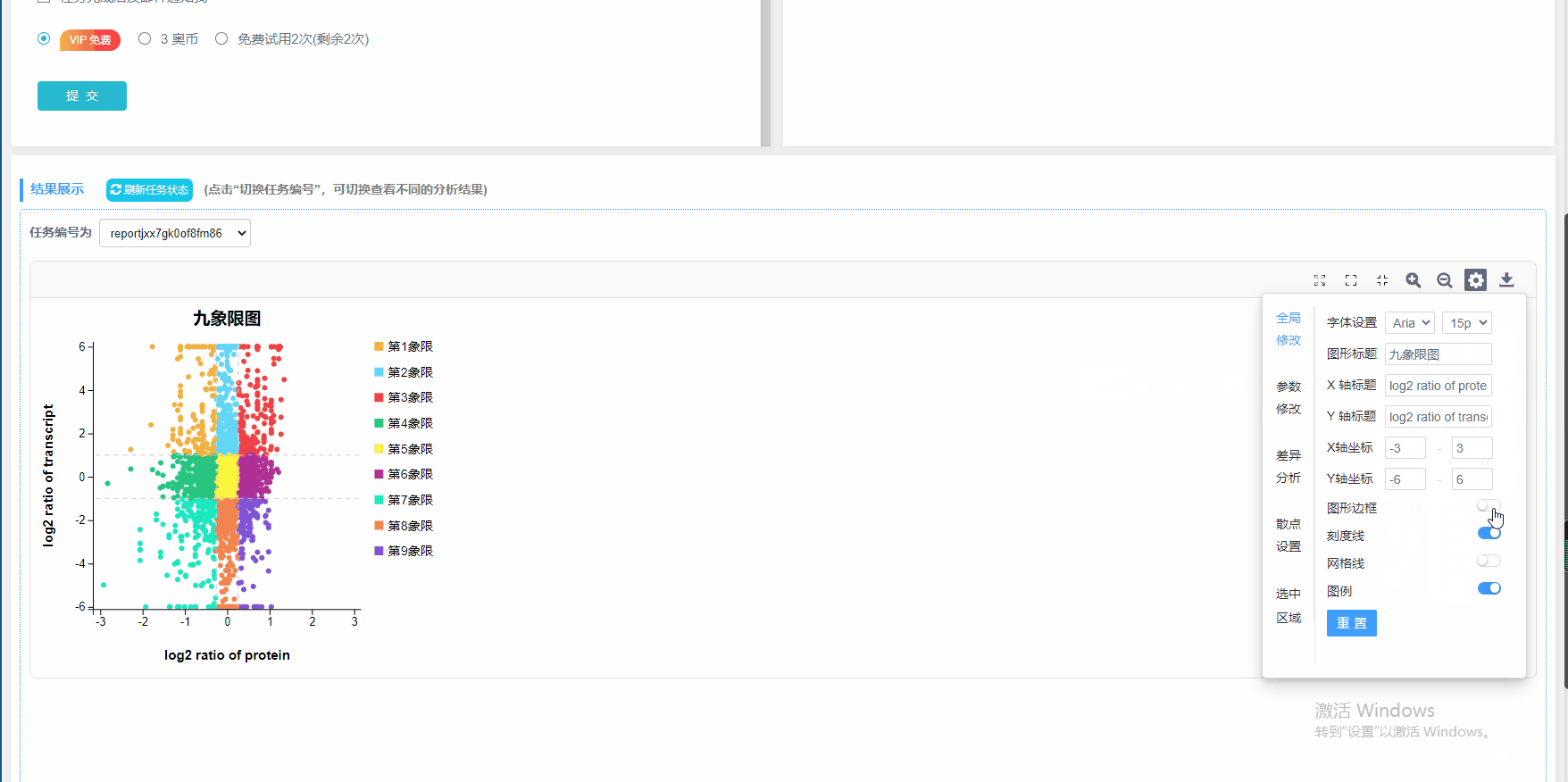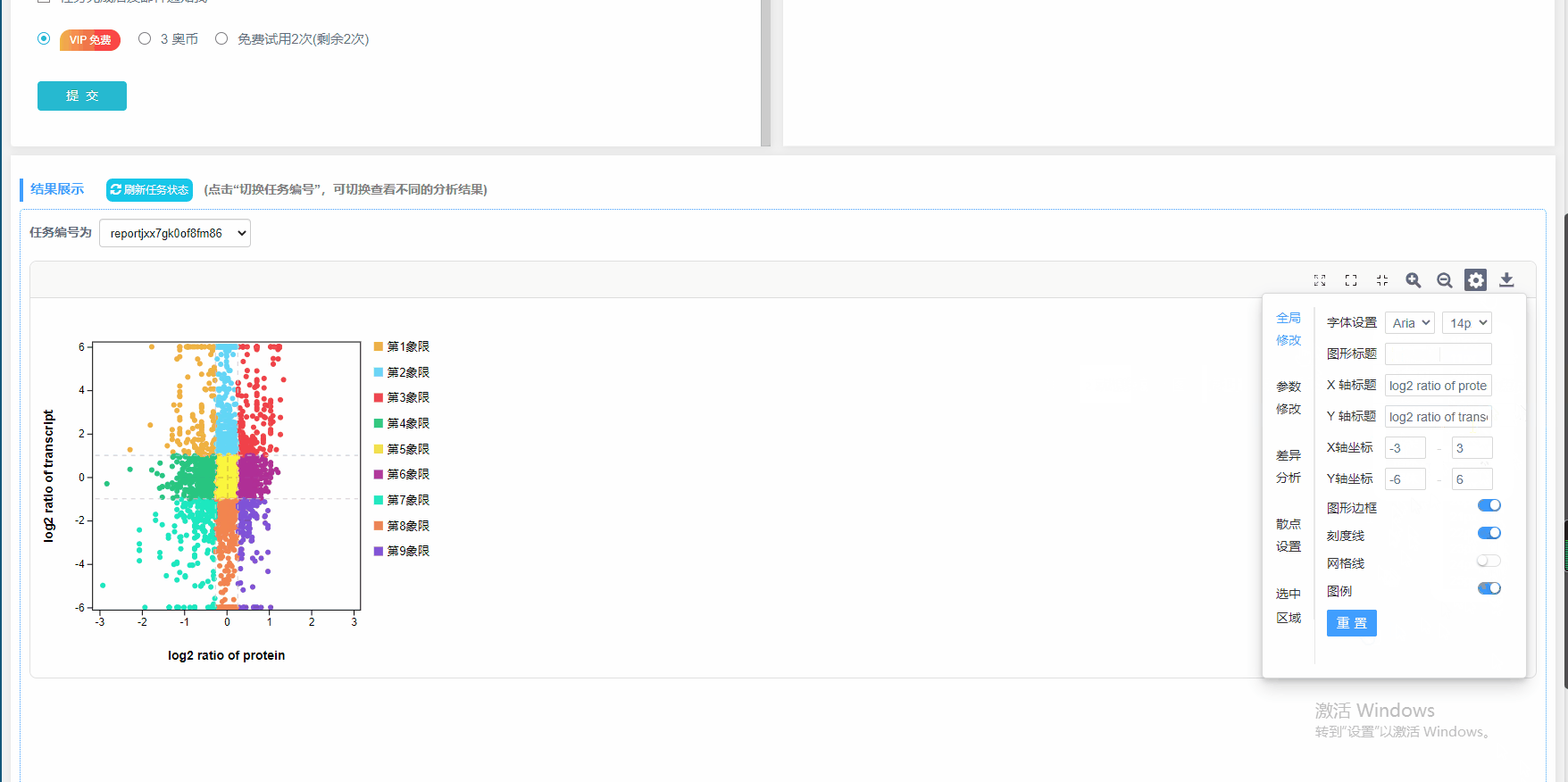
3)结果图:四/九象限图
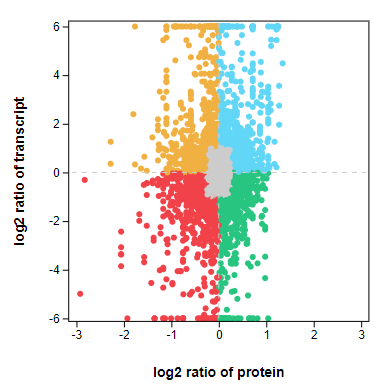
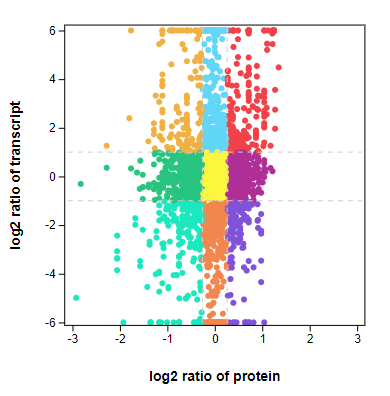
四象限图 九象限图
结果说明:
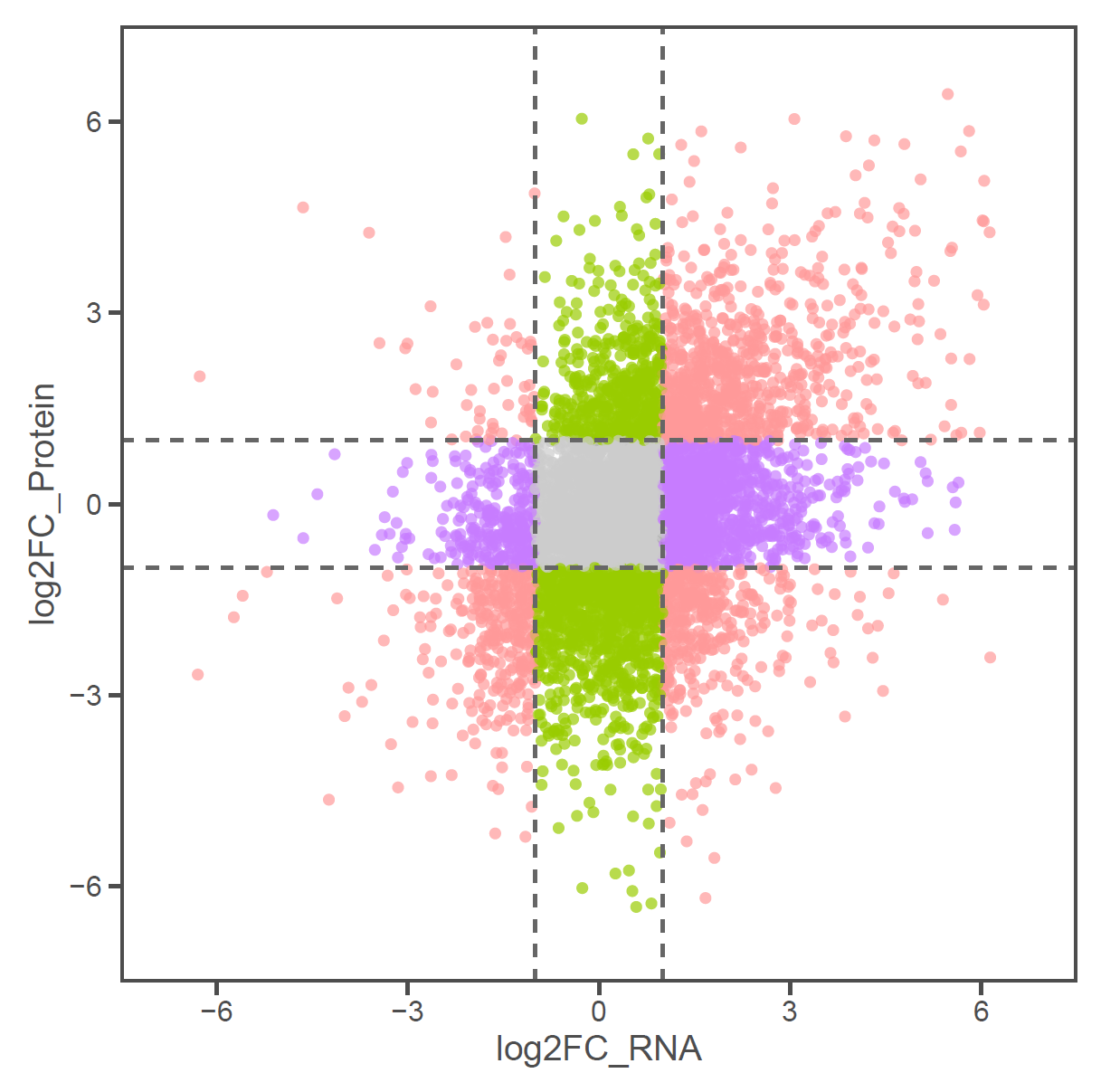
1)四象限图:每个点代表一个基因/蛋白,中心位置的点表示非差异的蛋白和基因,横轴上的点表示蛋白差异表达但基因非差异表达,纵轴上的点表示基因差异表达但蛋白非差异表达,1,4象限表示表达量负相关的基因/蛋白,2,3象限表示表达量正相关的基因/蛋白。
2)九象限图:每个点代表一个基因/蛋白,纵坐标代表转录组基因差异倍数的log2值,横坐标代表蛋白表达差异倍数的log2值。基因表达上下调2倍的基因为显著差异基因(log2=1或-1),蛋白表达上下调1.2倍的蛋白为显著差异蛋白(log2= 0.263,或- 0.263)
|
象限 |
解读 |
可挖掘的信息 |
|
1,9 |
mRNA与对应的蛋白差异表达模式负相关 |
转录后或翻译水平调控,例如miRNA调控靶基因导致抑制蛋白翻译 |
|
2,8 |
mRNA差异表达,对应蛋白无变化 |
转录后或翻译水平调控 |
|
3,7 |
mRNA与对应的蛋白差异表达模式正相关 |
转录和翻译水平同步变化的基因 |
|
4,6 |
蛋白差异表达,对应mRNA无变化 |
翻译水平调控或累积蛋白 |
|
5 |
共表达mRNA和蛋白均非差异表达 |
多数基因和蛋白非差异表达 |
Q1. 上传的数据需要保存成什么格式?文件名称和拓展名有没有要求?
OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。如果是核酸、蛋白序列文件,必须为FASTA格式(本质是文本文件)。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”“.fasta”等,例如 mydata01.txt,gene02.xlsx 。
Q2. 提交时报错常见问题:
1.提交时显示X行X列空行/无数据,请先自查表格中是否存在空格或空行,需要删掉。
2.提交时显示列数只有1列,但表格数据不止1列:列间需要用分隔符隔开,先行检查文件是否用了分隔符。
其它提示报错,请先自行根据提示修改;如果仍然无法提交,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q3. 提交的任务完成后却不出图该怎么办?
主要原因是上传的数据文件存在特殊符号所致。可参考以下建议逐一排查出错原因:
(1)数据中含中文字符,把中文改成英文;
(2) 数据中含特殊符号,例如 %、NA、+、-、()、空格、科学计数、罗马字母等,去掉特殊符号,将空值用数字“0”替换;
(3)检查数据中是否有空列、空行、重复的行、重复的列,特别是行名(一般为gene id)、列名(一般为样本名)出现重复值,如果有删掉。
排查完之后,重新上传数据、提交任务。如果仍然不出图,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q4.下载的结果文件用什么软件打开?
OmicShare云平台的结果文件(例如,下图为KEGG富集分析的结果文件)包括两种类型:图片文件和文本文件。
图片文件:
为了便于用户对图片进行后期编辑,OmicShare同时提供位图(png)和矢量图(pdf、svg)两种类型的图片。对于矢量图,最常见的是pdf和svg格式,常用Ai(Adobe illustrator)等进行编辑。其中,svg格式的图片可用网页浏览器打开,也可直接在word、ppt中使用。
文本文件:
文本文件的拓展名主要有4种类型:“.os”、“.xls”、“.log”和“.txt”。这些文件本质上都是制表符分隔的文本文件,使用记事本、Notepad++、EditPlus、Excel等文本编辑器直接打开即可。结果文件中,拓展名为“.os”文件为上传的原始数据;“.xls”文件一般为分析生成的数据表格;“.log”文件为任务运行日志文件,便于检查任务出错原因。
Q5. 提交的任务一直在排队怎么办?
提交任务后都需要排队,1分钟后,点击“任务状态刷新”按钮即可。除了可能需运行数天的注释工具,一般工具数十秒即可出结果,如果超出30分钟仍无结果,请联系OS客服,发送任务编号给OmicShare客服,会有专人为你处理任务问题。
Q6. 结果页面窗口有问题,图表加载不出来怎么办?
尝试用谷歌浏览器登录OmicShare查看结果文件,部分浏览器可能不兼容。
引用OmicShare Tools的参考文献为:
Mu, Hongyan, Jianzhou Chen, Wenjie Huang, Gui Huang, Meiying Deng, Shimiao Hong, Peng Ai, Chuan Gao, and Huangkai Zhou. 2024. “OmicShare tools: a Zero‐Code Interactive Online Platform for Biological Data Analysis and Visualization.” iMeta e228. https://doi.org/10.1002/imt2.228案例1:
发表期刊:Nature Communications
影响因子:16.6
发表时间:2023
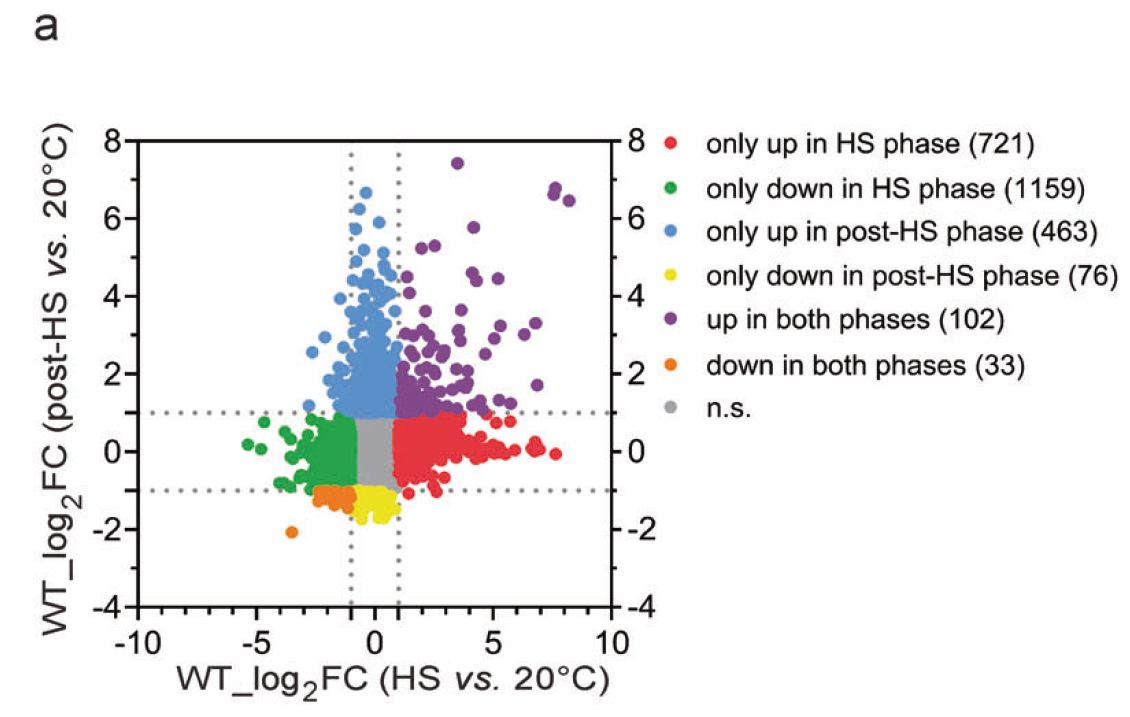
Fig. 2: Hormetic heat stress results in an ENDU-2-dependent reprogramming of the transcriptome after heat stress. a Scatter Plot comparing the average gene expression changes upon 1 h HS (x-axis) and 4 h after HS (y axis) in WT animals. Each dot corresponds to a gene. Genes showing with a significant increase or decrease in transcript level (FC > 2, FDR < 0.01) only upon 1 h HS are shaded red or green respectively.
引用方式:
The Graph was generated by OmicShare tool (https://www.omicshare.com).
参考文献:
Xu F, Li R, von Gromoff E D, et al. Reprogramming of the transcriptome after heat stress mediates heat hormesis in Caenorhabditis elegans[J]. Nature Communications, 2023, 14(1): 4176.
案例2:
发表期刊:Ecotoxicology and Environmental Safety
影响因子:6.8
发表时间:2023
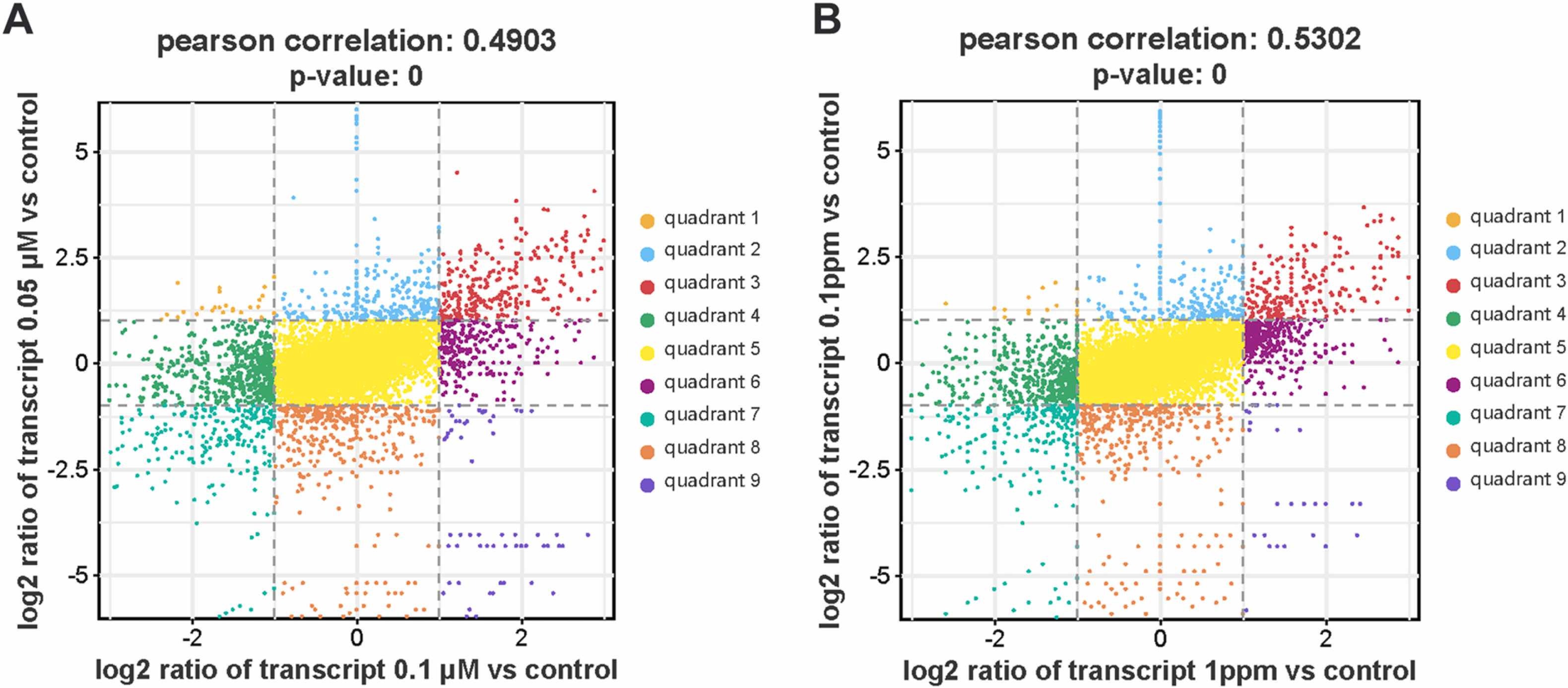
Fig. 3. The nine-quadrant map analysis of DEGs of experiment A and experiment B. The nine-quadrant map of experiment A was listed on the left (A), and the nine-quadrant map of experiment B was listed on the right (B). Different quadrants were represented by different colors, which have been marked in the picture. The dotted line was the threshold line for differential expression.
引用方式:
The genes which |fold change (log2)| > 1 were regarded as significantly differentially expressed in the nine-quadrant plot analysis. Nine-quadrant plot analysis was performed using OmicShare tools (https://www.omicshare.com/tools).
参考文献:
Yu H, Song W, Chen X, et al. Transcriptomic analysis reveals up-regulated histone genes may play a key role in zebrafish embryo-larvae response to Bisphenol A (BPA) exposure[J]. Ecotoxicology and Environmental Safety, 2023, 252: 114578.
案例3:
发表期刊:Infection and Drug Resistance
影响因子:3.9
发表时间:2022
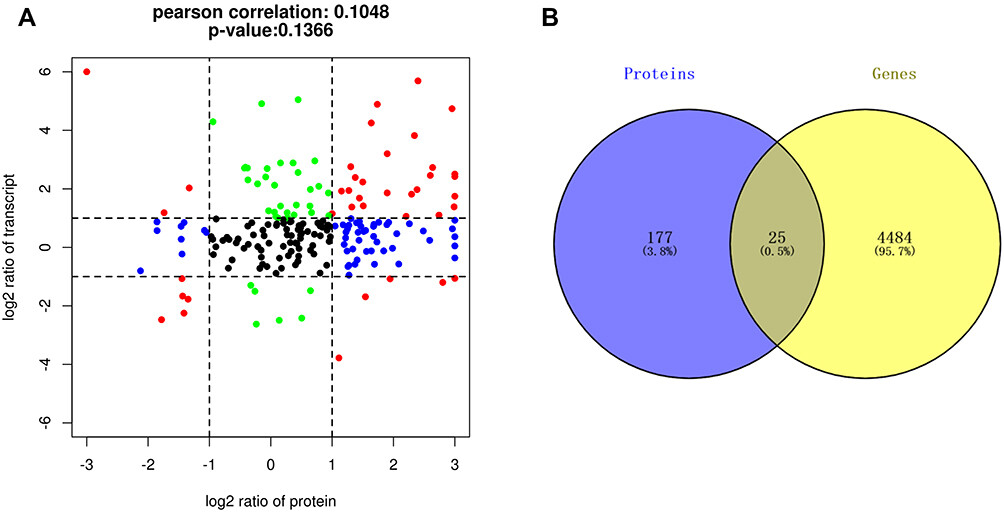
Figure 3 Transcriptome-proteome association analysis. (A) Nine-quadrant diagram showing 203 intersecting targets between transcriptome and proteome, including 8 targets in quadrants 1 or 9, 36 targets in quadrants 2 or 8, 31 targets in quadrants 3 or 7, 54 targets in quadrants 4 or 6, and 73 targets in quadrant 5.
引用方式:
Differentially expressed proteins were screened using the OmicShare online platform (https://www.omicshare.com/) with the screening criteria: p<0.05 and |FC|≥2.0. Transcriptomic and proteomic data are submitted to OmicShare platform to construct a nine-quadrant map.
参考文献:
Wang C, Li Y, Li S, et al. Proteomics combined with RNA sequencing to screen biomarkers of sepsis[J]. Infection and Drug Resistance, 2022: 5575-5587.
案例4:
发表期刊:The Plant Journal
影响因子:7.2
发表时间:2020
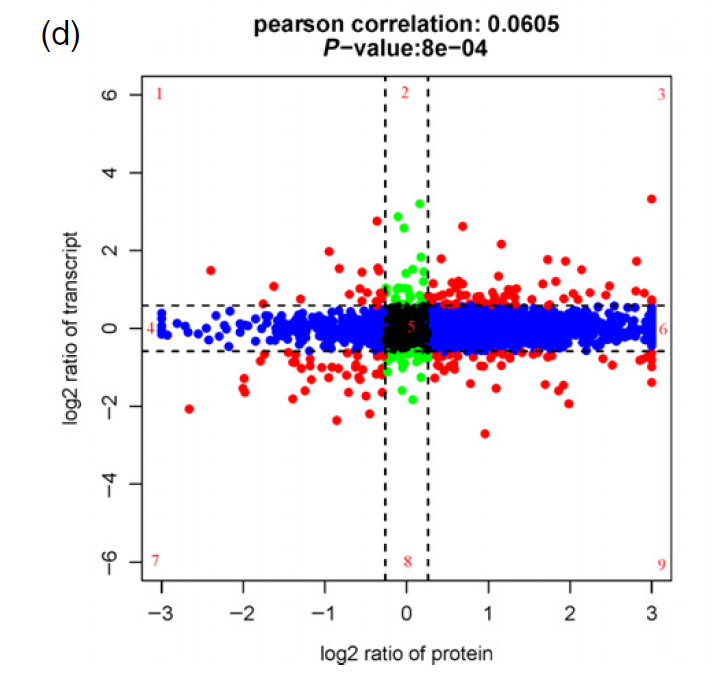
Fig 9. Correlation between protein expression and mRNA expression during grain filling under soil drying conditions. (d) Correlated proteins and RNAs enriched in nine quadrants at 24 DAA. Quadrants 1, 2 and 4 indicate that the protein abundance was lower than the RNA abundance. In quadrants 3 and 7, the RNAs correspond with the related proteins. Quadrant 5 shows that the proteins and RNAs were commonly expressed with no difference. Quadrants 6, 8 and 9 indicate that the protein abundance was higher than the RNA abundance.
引用方式:
Furthermore, we performed a nine-quadrant analysis via the omicshare platform (http://www.omicshare.com/tools/Home/Soft/jxx) to examine the relationship between the proteins and RNAs detected under soil drying treatment.
参考文献:
Wang G, Li H, Wang K, et al. Regulation of gene expression involved in the remobilization of rice straw carbon reserves results from moderate soil drying during grain filling[J]. The Plant Journal, 2020, 101(3): 604-618.





 扫码支付更轻松
扫码支付更轻松



