1. 全局修改
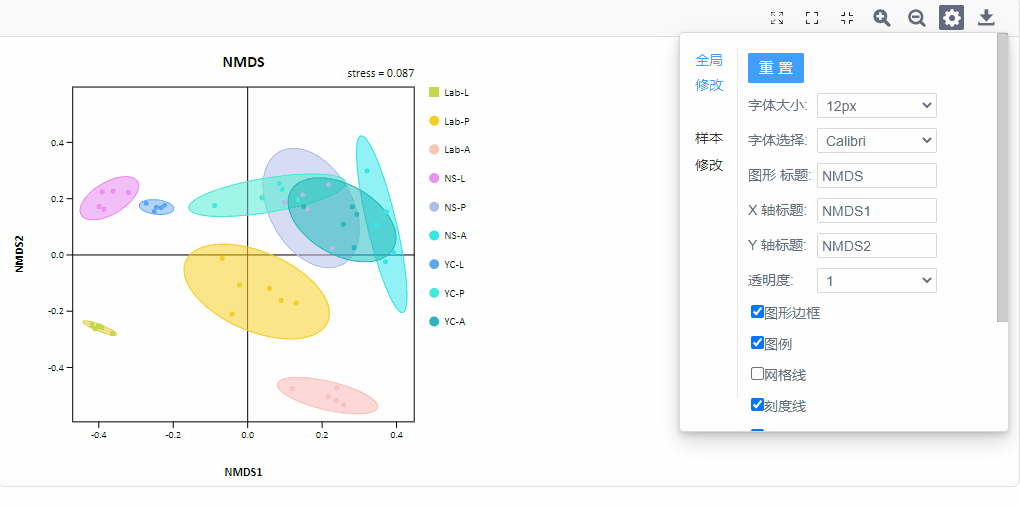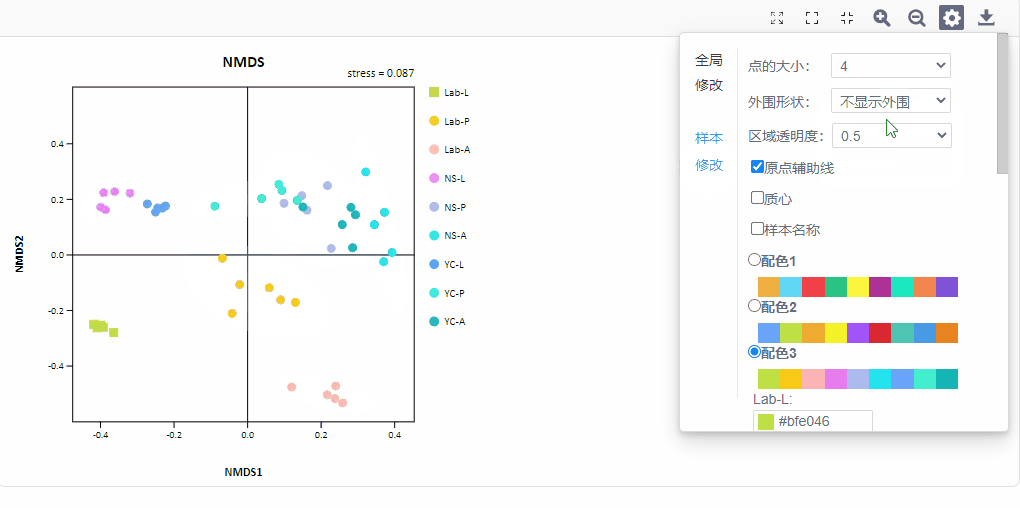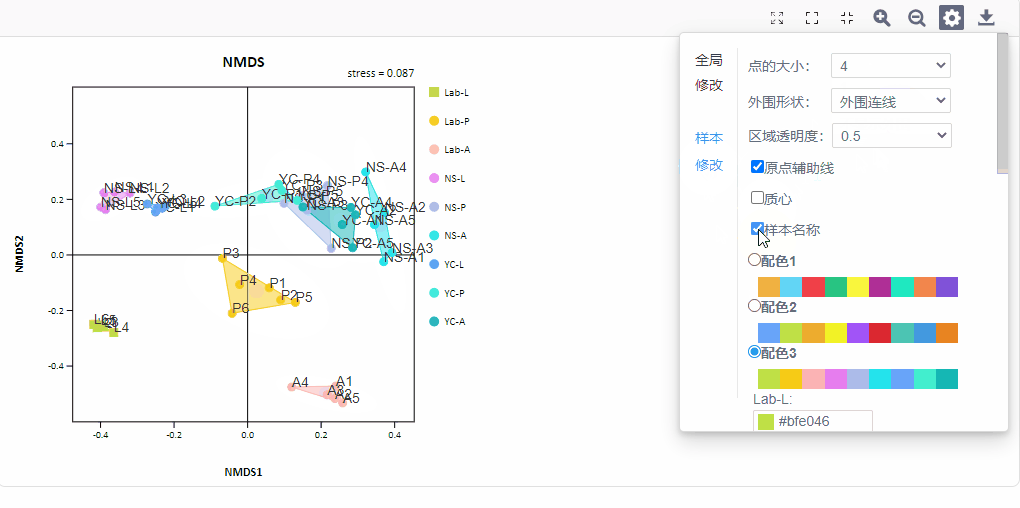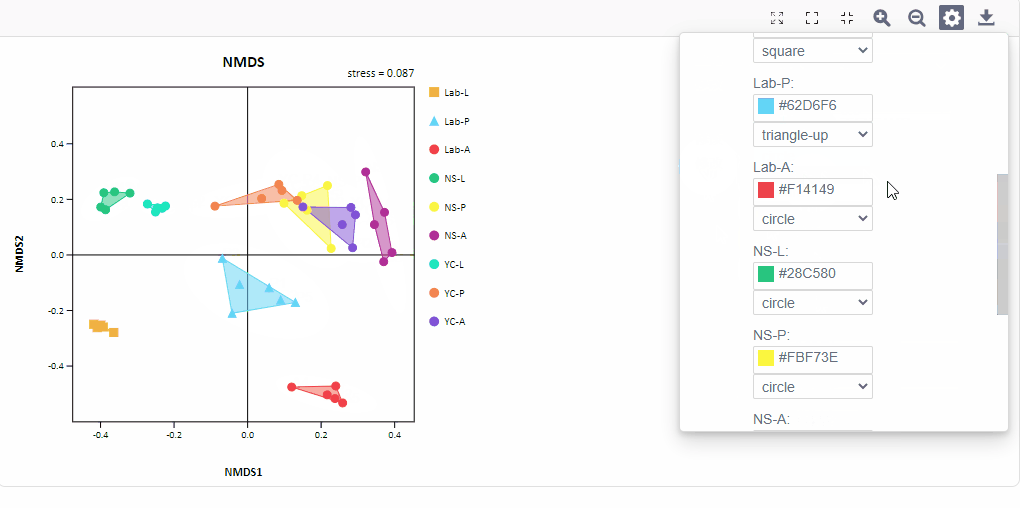
2. 样本修改
3. 图大小调整和下载
1.功能
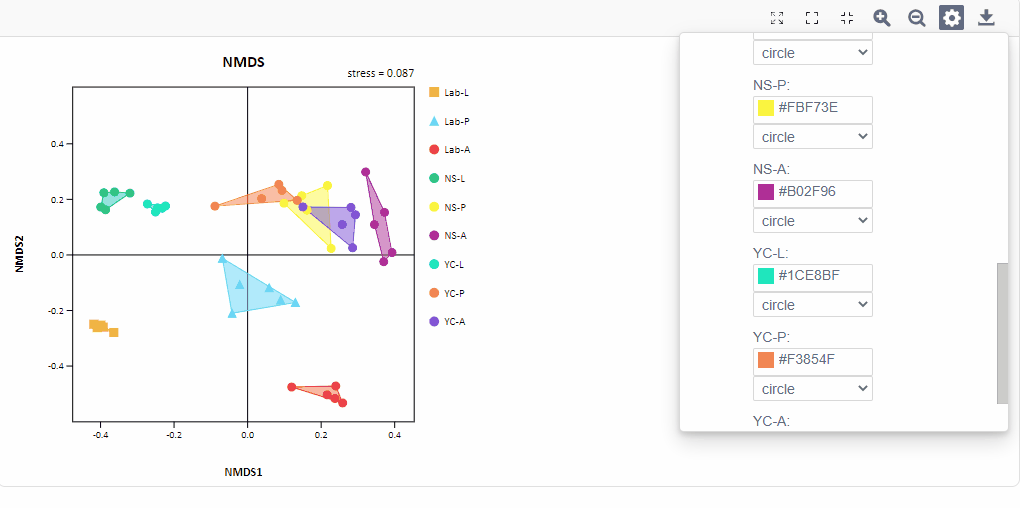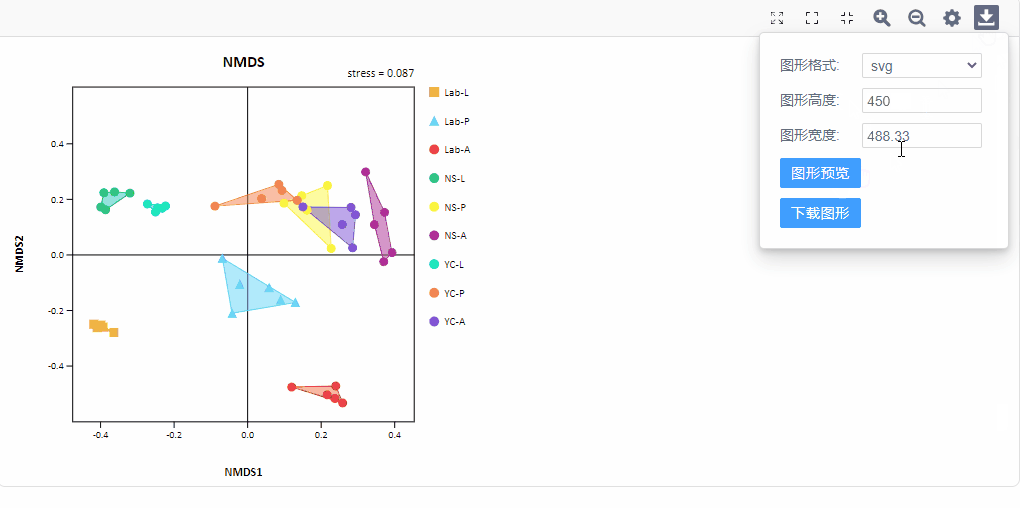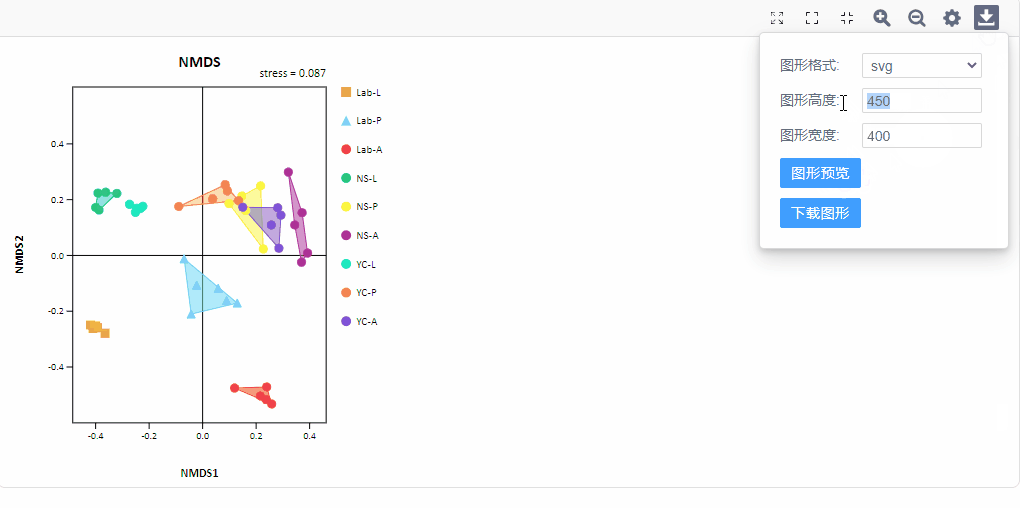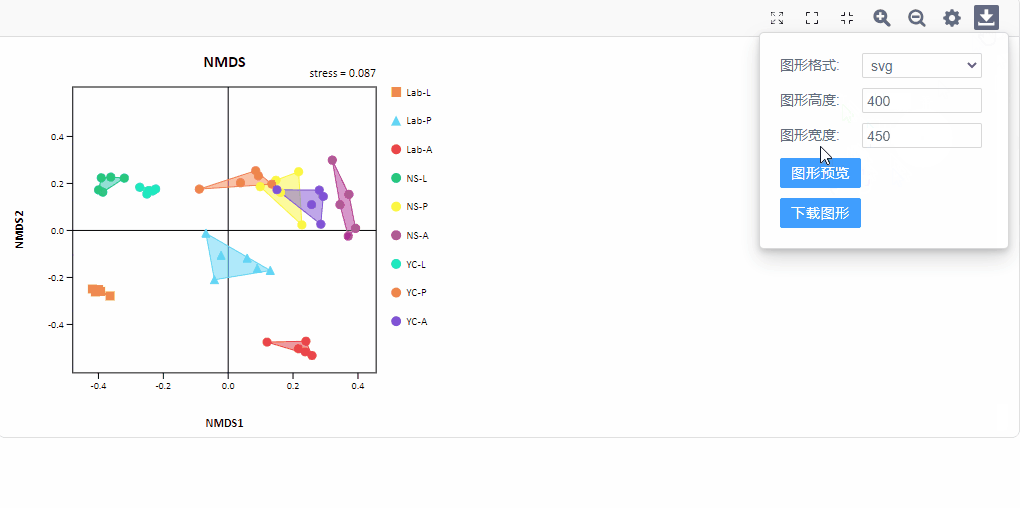
基于物种丰度表或样本间距离矩阵,进行NMDS(non-metric multi-dimensional scaling, NMDS)分析并动态展示。常用于直观呈现样本与分组的聚类特征。
备注:若输入丰度表,默认基于Bray-Curtis距离进行分析。
2.应用范围
16S、宏基因组等测序分析的丰度表/距离表。
3.输入
1)文件格式:支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”等,例如 mydata01.txt,gene02.xlsx 。
2)必须:丰度表或距离矩阵;非必须:样本分组文件。
4.分析操作
1)坐标轴刻度范围自适应或自定义
2)修改样本点颜色、形状、大小、透明度
3)添加样本95%置信区间的椭圆或添加组内样本间外围连线
4)是否显示原点辅助线、样本名称、质心
5)是否显示stress值
6)在任务编号右侧打包下载绘图数据
5.图形解读
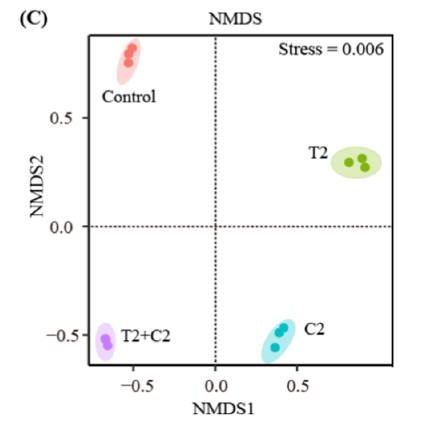
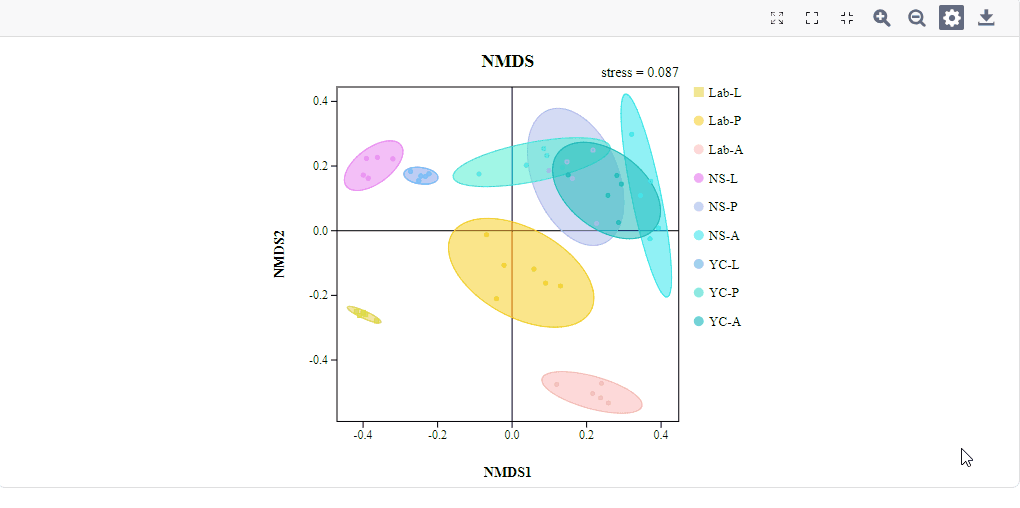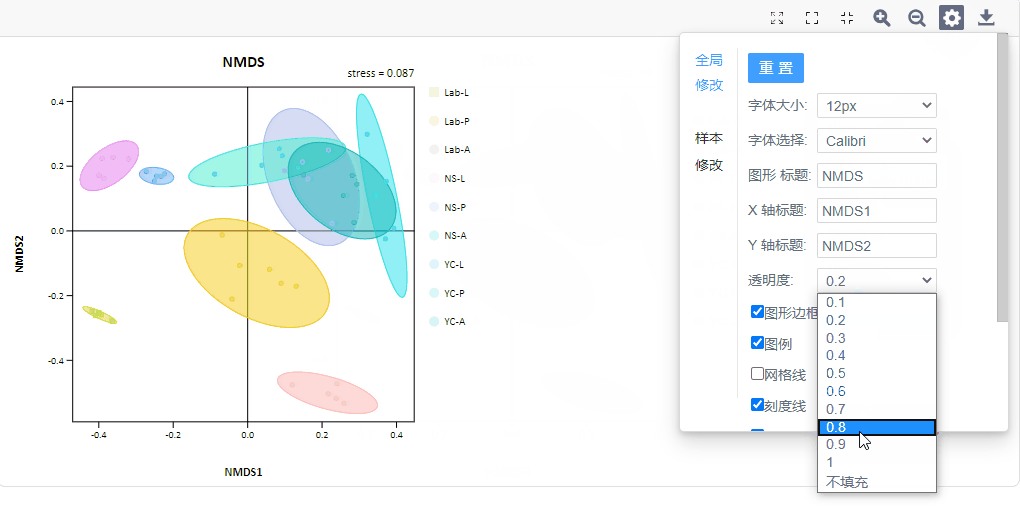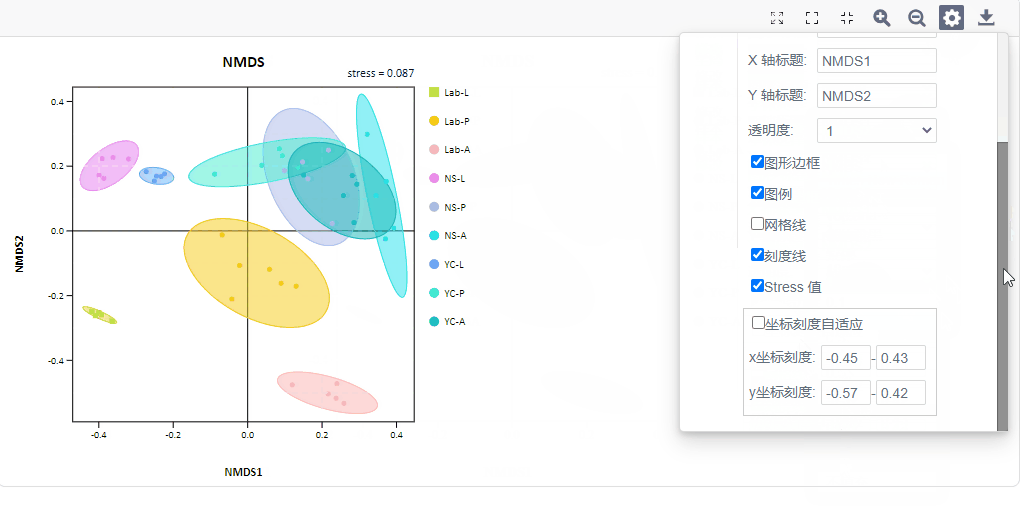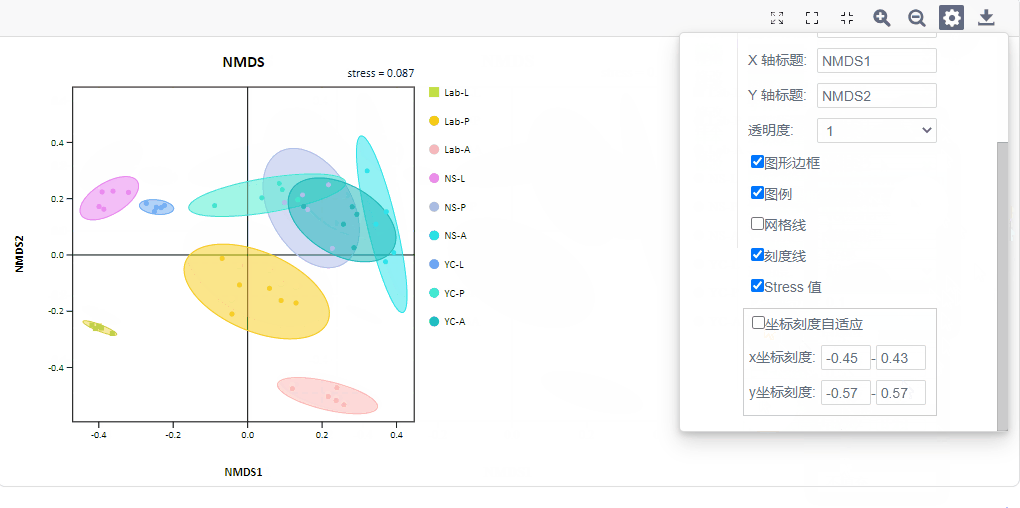
如下图,图中点表示样本,以样本在二维平面的距离关系反映样本间菌群结构的相似性,样本距离越近,说明样本间的物种组成越相似。stress值表示模型效果,值越小,模型越可靠,一般要求stress值至少小于0.1。

Q1. 上传的数据需要保存成什么格式?文件名称和拓展名有没有要求?
OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。如果是核酸、蛋白序列文件,必须为FASTA格式(本质是文本文件)。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”“.fasta”等,例如 mydata01.txt,gene02.xlsx 。
Q2. 提交时报错常见问题:
1.提交时显示X行X列空行/无数据,请先自查表格中是否存在空格或空行,需要删掉。
2.提交时显示列数只有1列,但表格数据不止1列:列间需要用分隔符隔开,先行检查文件是否用了分隔符。
其它提示报错,请先自行根据提示修改;如果仍然无法提交,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q3. 提交的任务完成后却不出图该怎么办?
主要原因是上传的数据文件存在特殊符号所致。可参考以下建议逐一排查出错原因:
(1)数据中含中文字符,把中文改成英文;
(2) 数据中含特殊符号,例如 %、NA、+、-、()、空格、科学计数、罗马字母等,去掉特殊符号,将空值用数字“0”替换;
(3)检查数据中是否有空列、空行、重复的行、重复的列,特别是行名(一般为gene id)、列名(一般为样本名)出现重复值,如果有删掉。
排查完之后,重新上传数据、提交任务。如果仍然不出图,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q4.下载的结果文件用什么软件打开?
OmicShare云平台的结果文件(例如,下图为KEGG富集分析的结果文件)包括两种类型:图片文件和文本文件。
图片文件:
为了便于用户对图片进行后期编辑,OmicShare同时提供位图(png)和矢量图(pdf、svg)两种类型的图片。对于矢量图,最常见的是pdf和svg格式,常用Ai(Adobe illustrator)等进行编辑。其中,svg格式的图片可用网页浏览器打开,也可直接在word、ppt中使用。
文本文件:
文本文件的拓展名主要有4种类型:“.os”、“.xls”、“.log”和“.txt”。这些文件本质上都是制表符分隔的文本文件,使用记事本、Notepad++、EditPlus、Excel等文本编辑器直接打开即可。结果文件中,拓展名为“.os”文件为上传的原始数据;“.xls”文件一般为分析生成的数据表格;“.log”文件为任务运行日志文件,便于检查任务出错原因。
Q5. 提交的任务一直在排队怎么办?
提交任务后都需要排队,1分钟后,点击“任务状态刷新”按钮即可。除了可能需运行数天的注释工具,一般工具数十秒即可出结果,如果超出30分钟仍无结果,请联系OS客服,发送任务编号给OmicShare客服,会有专人为你处理任务问题。
Q6. 结果页面窗口有问题,图表加载不出来怎么办?
尝试用谷歌浏览器登录OmicShare查看结果文件,部分浏览器可能不兼容。
引用OmicShare Tools的参考文献为:
Mu, Hongyan, Jianzhou Chen, Wenjie Huang, Gui Huang, Meiying Deng, Shimiao Hong, Peng Ai, Chuan Gao, and Huangkai Zhou. 2024. “OmicShare tools: a Zero‐Code Interactive Online Platform for Biological Data Analysis and Visualization.” iMeta e228. https://doi.org/10.1002/imt2.228案例1:
发表期刊:Environmental Science & Technology
影响因子:11.4
发表时间:2023
Figure 2. Microbial composition (A), and the intergroup difference (C) in seawater after 60 days of HTO, C-14 exposure.
引用方式:The OmicShare platform was used for clustering heatmaps, NMDS, and RDA analysis (https://www.omicshare.com/tools/).
参考文献:
Lai J, Li Z, Wang Y, et al. Tritium and Carbon-14 Contamination Reshaping the Microbial Community Structure, Metabolic Network, and Element Cycle in the Seawater Environment[J]. Environmental Science & Technology, 2023, 57(13): 5305-5316.
案例2:
发表期刊:Applied Biochemistry and Biotechnology
影响因子:3.0
发表时间:2022
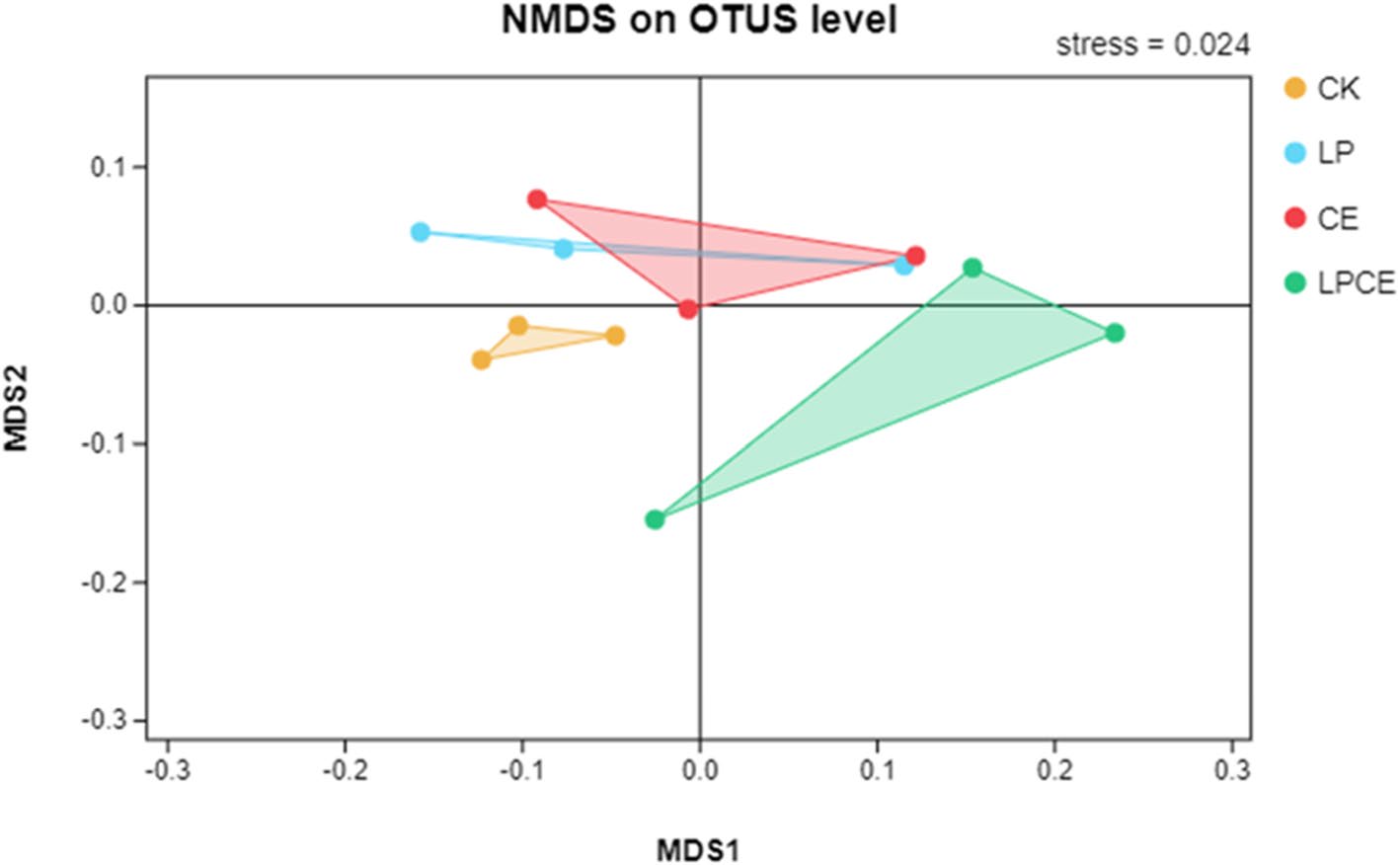
Fig 4. Differences in relative abundance of microbial community on OTU level shown by non-metric multidimensional scaling (NMDS)
引用方式:High-throughput sequencing data were analyzed using the OmicShare tools (https:// www. omics hare. com/ tools).
参考文献:
Wang Q, Wang R, Wang C, et al. Effects of cellulase and Lactobacillus plantarum on fermentation quality, chemical composition, and microbial community of mixed silage of whole-plant corn and peanut vines[J]. Applied Biochemistry and Biotechnology, 2022, 194(6): 2465-2480.
案例3:
发表期刊:Water Research
影响因子:12.8
发表时间:2020
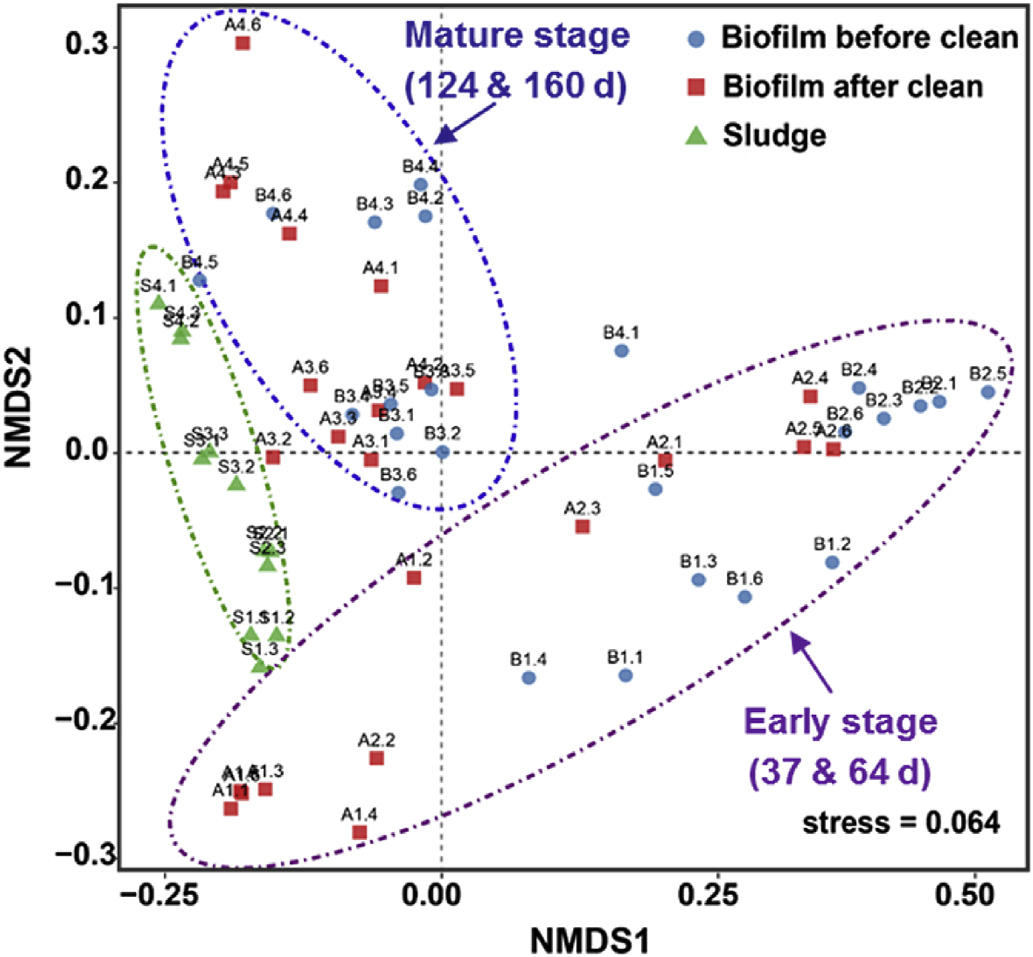
Fig. 3. Nonmetric multidimensional scaling (NMDS) analysis of membrane biofilm and sludge samples based on weighted Uni-Frac distance. The numbers from 1 to 4 refer to the sampling time of the 37th, 64th, 124th and 160th days.
引用方式:The beta diversity was analyzed based on weighted Unifracdistance calculating the total OTUs (Chang et al., 2011) and visualized by non-metric multi-dimensional scaling (NMDS) using OmicShare tools (http://www.omicshare.com/tools).
参考文献:
Wang H, Liu Z, Luo S, et al. Membrane autopsy deciphering keystone microorganisms stubborn against online NaOCl cleaning in a full-scale MBR[J]. Water Research, 2020, 171: 115390.





 扫码支付更轻松
扫码支付更轻松