一.功能
生存分析(Survival Analysis),结合终点事件出现与否和到达终点所经历时间的一类统计分析方法。
使用Kaplan-Meier法(极限乘积法,非参数估计),计算每个时间节点的生存概率,并根据概率乘法原理计算生存率,最后将每个时间节点所对应的生存率连接成生存曲线,观测在生存时间内终点事件的发生状况。对于多组生存资料,使用Logrank检验进行多条生存曲线间的差异比较。

二.适用范围
广泛适用于疾病、癌症的发生、发展或预后研究等情景,如探究不同因素如癌症分期、种族、性别、是否吸烟、不同治疗方案、风险评分高低、淋巴结转移情况、基因表达量高低等在生存时间内对患者生死的影响。
三.输入
文件格式:支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。
数据整理:长数据格式,共四列,第一行为列名
1. 第1列(行名列):如样本名、id编号等,不能出现重复值;
2. 第2列(终点事件列):为二分类变量0或1,1对应终点事件结局(如患者死亡,疾病痊愈等);
3. 第3列(生存时间列):如总生存期、无病生存期、无进展生存期等等。数值支持小数点,如200天可以是200/6.67/0.55(天/月/年);
4. 第4列(分组信息列):分类型数据,如男/女、抽烟/不抽烟。若想要探究的数据为连续型变量,如年龄、基因表达量、风险评分等等,需自行划分分组,如根据中位数将目标基因表达量拆分为高表达、低表达两组,将风险评分拆为高风险、低风险两组,将年龄拆分为幼年、青年、中年、老年等。
范例数据:
1)分组支持字符型数据
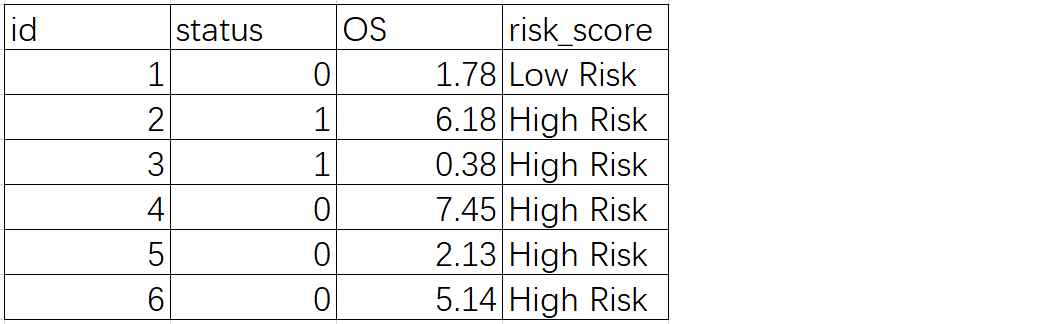
2)分组支持数字/罗马数字数据
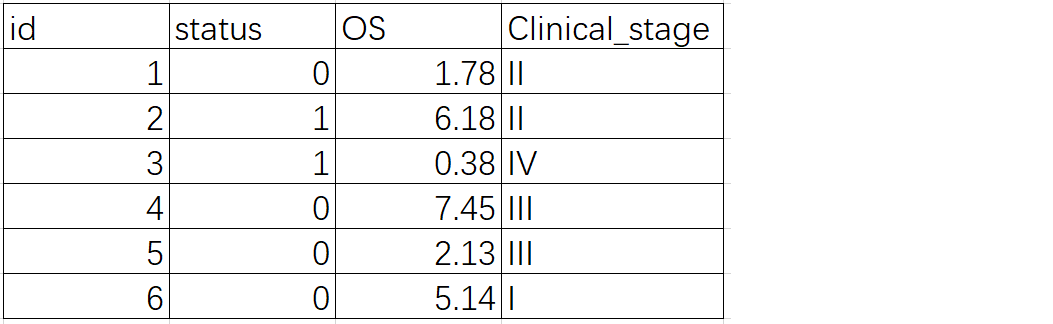
四.参数说明
1. 根据自身需求选择是否需要在生存分析主图中显示删失点、置信区间、P值和中位生存时间线;
2. 风险表和删失数据表会在结果文件中单独输出,选择显示/隐藏,仅影响生存分析主图效果(组合图拼接);
3. 添加颜色数和分组数一致,颜色顺序默认与分组字符首字母顺序一致(从小到大)。如Low和High两个分组,添加的第一个颜色对应High;I—IV四个分组,添加的第一个颜色对应I。可选择颜色添加,也可自行输入十六进制颜色码(英文逗号分隔)。
五.主图效果参考
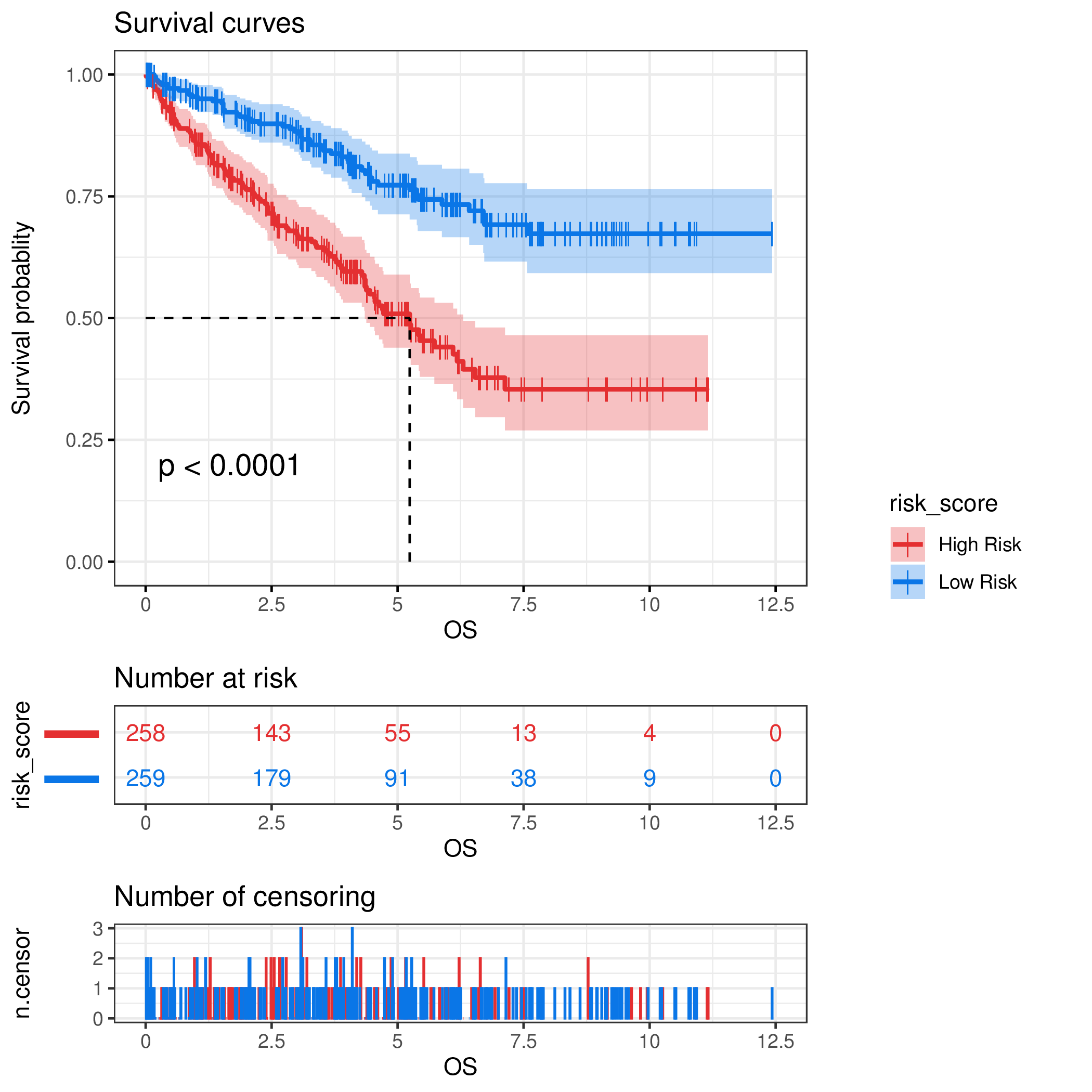
1)显示风险表/删失数据表
2)显示风险表+隐藏删失数据表

3)隐藏风险表/删失数据表+隐藏置信区间
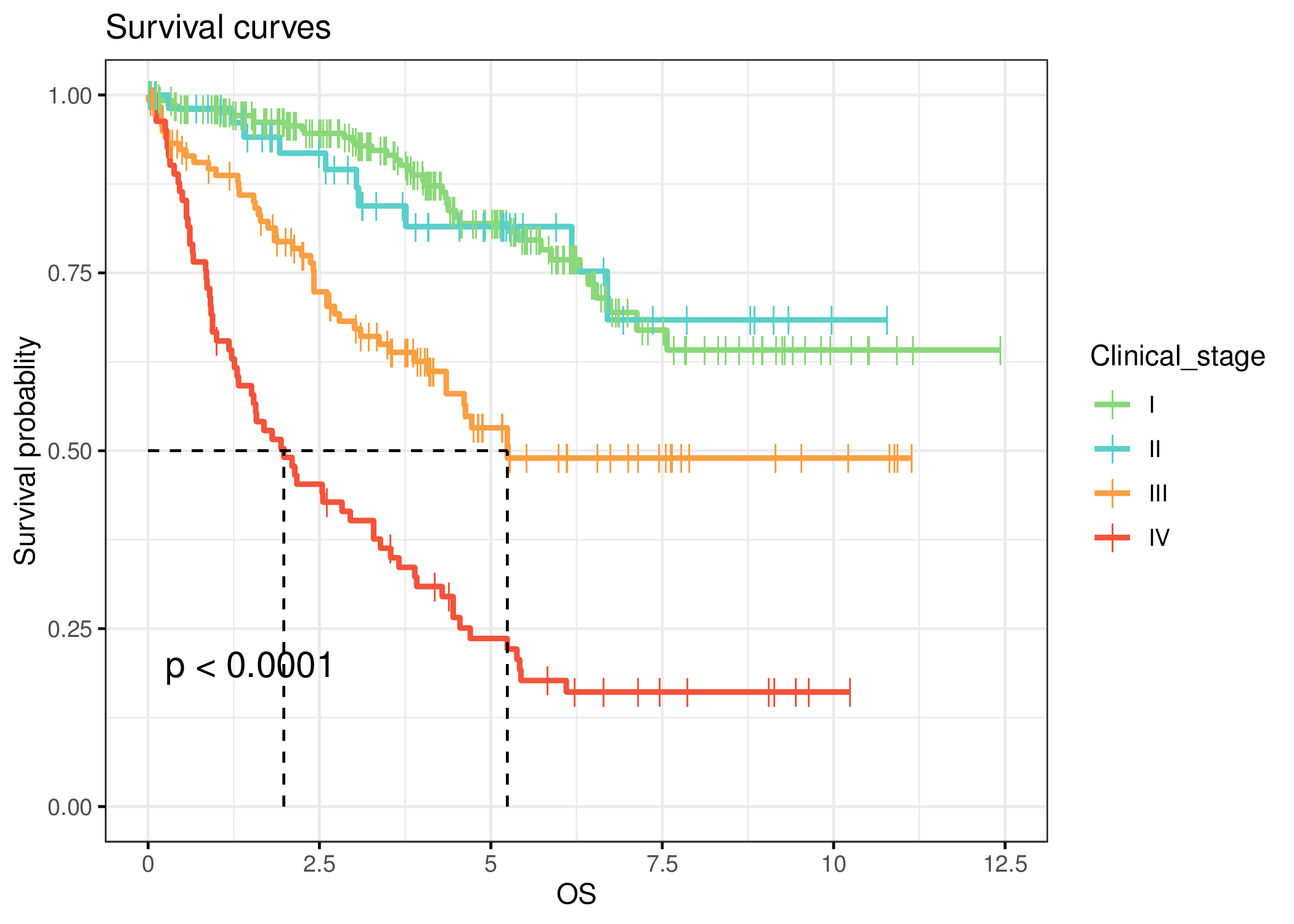
六.结果输出
1) surv.png/pdf:生存分析主图
2) risk.png/pdf:风险表
3) ncensor.png/pdf:删失数据表
4) survdiff.xls:差异检验结果表
结果表返回内容:
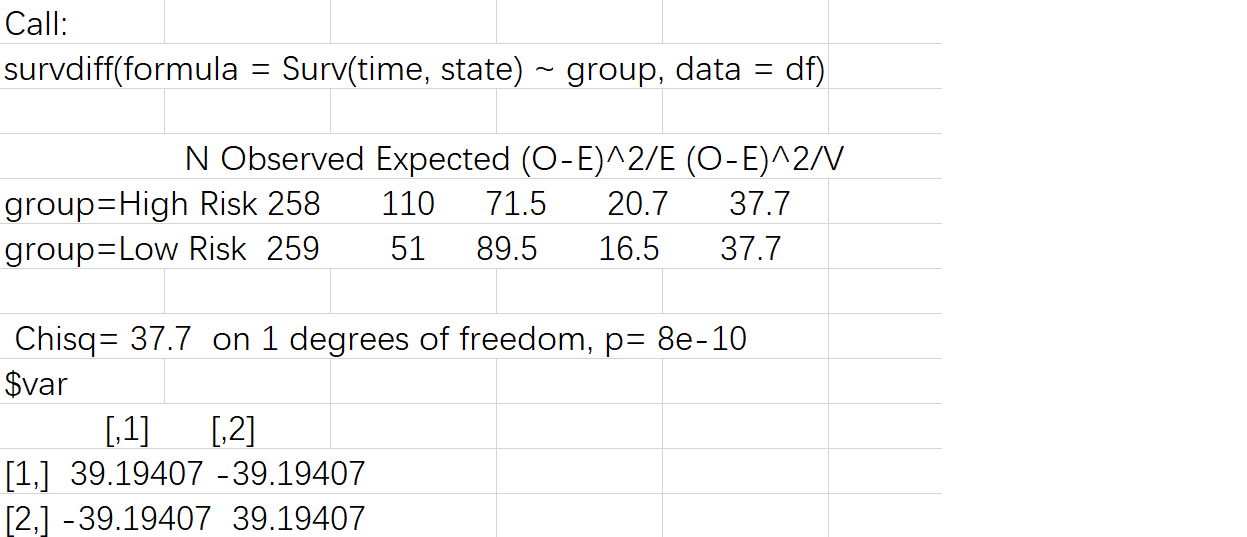
N:每组样本数
Observed:每组观测到的加权事件数
Expected:每组加权预期事件数
Chisq:卡方统计量
Var:方差矩阵
P:P值
绘图R包:survival + survminer
一. 数据整理与上传
通过“说明”和“示例文件”两处查看数据格式,按要求整理。点击“选择文件”上传整理好的数据。
二. 参数选择与提交
根据自身需求进行参数选择,详见“说明”部分,最后提交任务即可。

三. 结果查看
在“我的项目”中可查看和下载分析结果。
本地文件包含:
1) surv.png/pdf:生存分析主图
2) risk.png/pdf:风险表
3) ncensor.png/pdf:删失数据表
4) survdiff.xls:差异检验结果表
四. 结果解读

1) Survival curves(生存曲线):X轴为生存时间,Y轴为累积生存率,反映了不同分组间研究对象生存率随时间的变化。如上图,高风险组患者的生存率显著低于低风险组;生存曲线中短竖线为删失点,可以在参数选择中隐藏 。
2) Number at risk(风险表):风险表展示不同时间节点中有发生结局事件风险的所有研究对象的数量(即研究对象还未发生结局事件,也没有删失)。
3) Number of censoring(删失表):若在研究中未观察到研究对象出现结局事件,则标记为删失。删失表反映了不同时间点对应的研究对象删失数量,和生存曲线中的删失点一一对应。
Q1. 上传的数据需要保存成什么格式?文件名称和拓展名有没有要求?
OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。如果是核酸、蛋白序列文件,必须为FASTA格式(本质是文本文件)。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”“.fasta”等,例如 mydata01.txt,gene02.xlsx 。
Q2. 提交时报错常见问题:
1.提交时显示X行X列空行/无数据,请先自查表格中是否存在空格或空行,需要删掉。
2.提交时显示列数只有1列,但表格数据不止1列:列间需要用分隔符隔开,先行检查文件是否用了分隔符。
其它提示报错,请先自行根据提示修改;如果仍然无法提交,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q3. 提交的任务完成后却不出图该怎么办?
主要原因是上传的数据文件存在特殊符号所致。可参考以下建议逐一排查出错原因:
(1)数据中含中文字符,把中文改成英文;
(2) 数据中含特殊符号,例如 %、NA、+、-、()、空格、科学计数、罗马字母等,去掉特殊符号,将空值用数字“0”替换;
(3)检查数据中是否有空列、空行、重复的行、重复的列,特别是行名(一般为gene id)、列名(一般为样本名)出现重复值,如果有删掉。
排查完之后,重新上传数据、提交任务。如果仍然不出图,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q4.下载的结果文件用什么软件打开?
OmicShare云平台的结果文件(例如,下图为KEGG富集分析的结果文件)包括两种类型:图片文件和文本文件。
图片文件:
为了便于用户对图片进行后期编辑,OmicShare同时提供位图(png)和矢量图(pdf、svg)两种类型的图片。对于矢量图,最常见的是pdf和svg格式,常用Ai(Adobe illustrator)等进行编辑。其中,svg格式的图片可用网页浏览器打开,也可直接在word、ppt中使用。
文本文件:
文本文件的拓展名主要有4种类型:“.os”、“.xls”、“.log”和“.txt”。这些文件本质上都是制表符分隔的文本文件,使用记事本、Notepad++、EditPlus、Excel等文本编辑器直接打开即可。结果文件中,拓展名为“.os”文件为上传的原始数据;“.xls”文件一般为分析生成的数据表格;“.log”文件为任务运行日志文件,便于检查任务出错原因。
Q5. 提交的任务一直在排队怎么办?
提交任务后都需要排队,1分钟后,点击“任务状态刷新”按钮即可。除了可能需运行数天的注释工具,一般工具数十秒即可出结果,如果超出30分钟仍无结果,请联系OS客服,发送任务编号给OmicShare客服,会有专人为你处理任务问题。
Q6. 结果页面窗口有问题,图表加载不出来怎么办?
尝试用谷歌浏览器登录OmicShare查看结果文件,部分浏览器可能不兼容。





 扫码支付更轻松
扫码支付更轻松



