1.功能
序列LOGO工具,用于绘制核酸序列或蛋白序列的LOGO图,绘图效果如下。
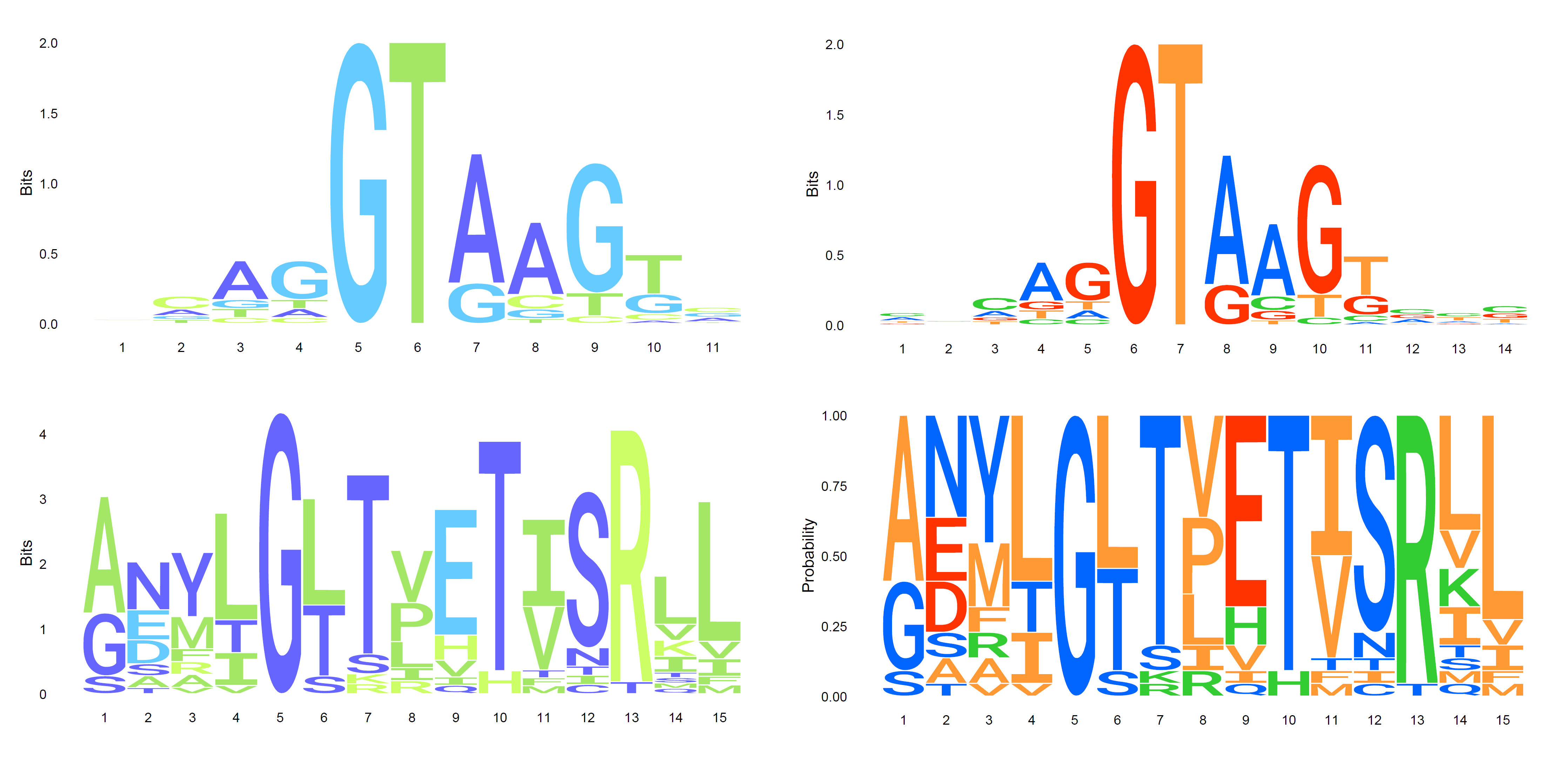
2.应用范围
分析和展示蛋白(或核酸)序列motif的保守性。
3.输入文件
支持两种格式的蛋白或核酸序列文件,本质上都是TXT文本文件。
a. FASTA格式的对位序列文件,序列名称用符号>标记,且序列的长度必须相同;
b. TXT格式的对位序列文件,每一行表示一条序列,且序列的长度必须相同。

4.主要参数
忽略小写字母(Ignore lower case): 是否忽略序列中的小写字母,默认是,即小写字母不参与计算展示;
单位(Units): 有bits 和 probability 供用户选择,默认bits。LOGO图纵坐标的单位常见有两种,一种是百分比,另一种是bits。对于probability,很好理解,每个字母的出现频率;对于bits,可参考下面的公式:
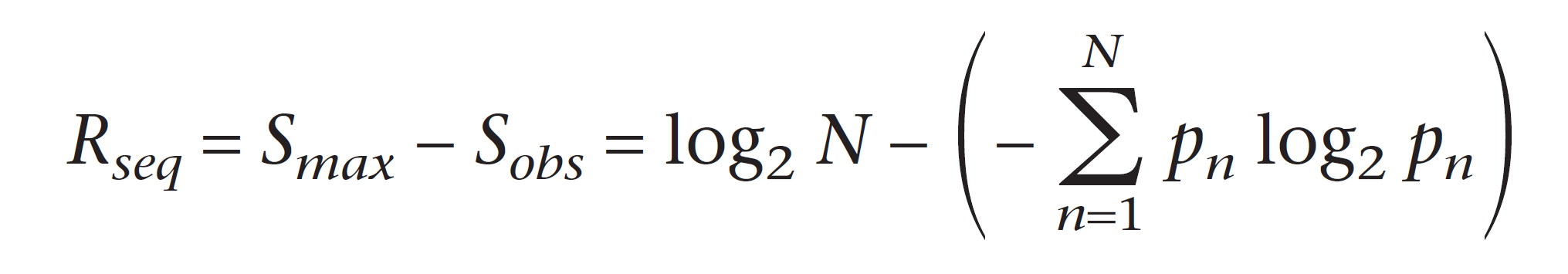
注:p n是相应位置n上相应字符出现频率,N是不同字符的总数量(核酸为4,蛋白质为20)。因此,对于图中的y轴的最大数值就不难理解,核酸序列的最大值为log2 4 = 2bits,蛋白序列为log2 20≈4.32 bits。
首字母编号(First position number): 默认从1开始;
LOGO范围(Logo range): 指定显示序列范围,默认logo start-logo end,即显示全部序列;
配色方案(Color scheme): 默认使用自动配色(Auto),也可选择自定义配色(Custom)方案。
5.结果输出
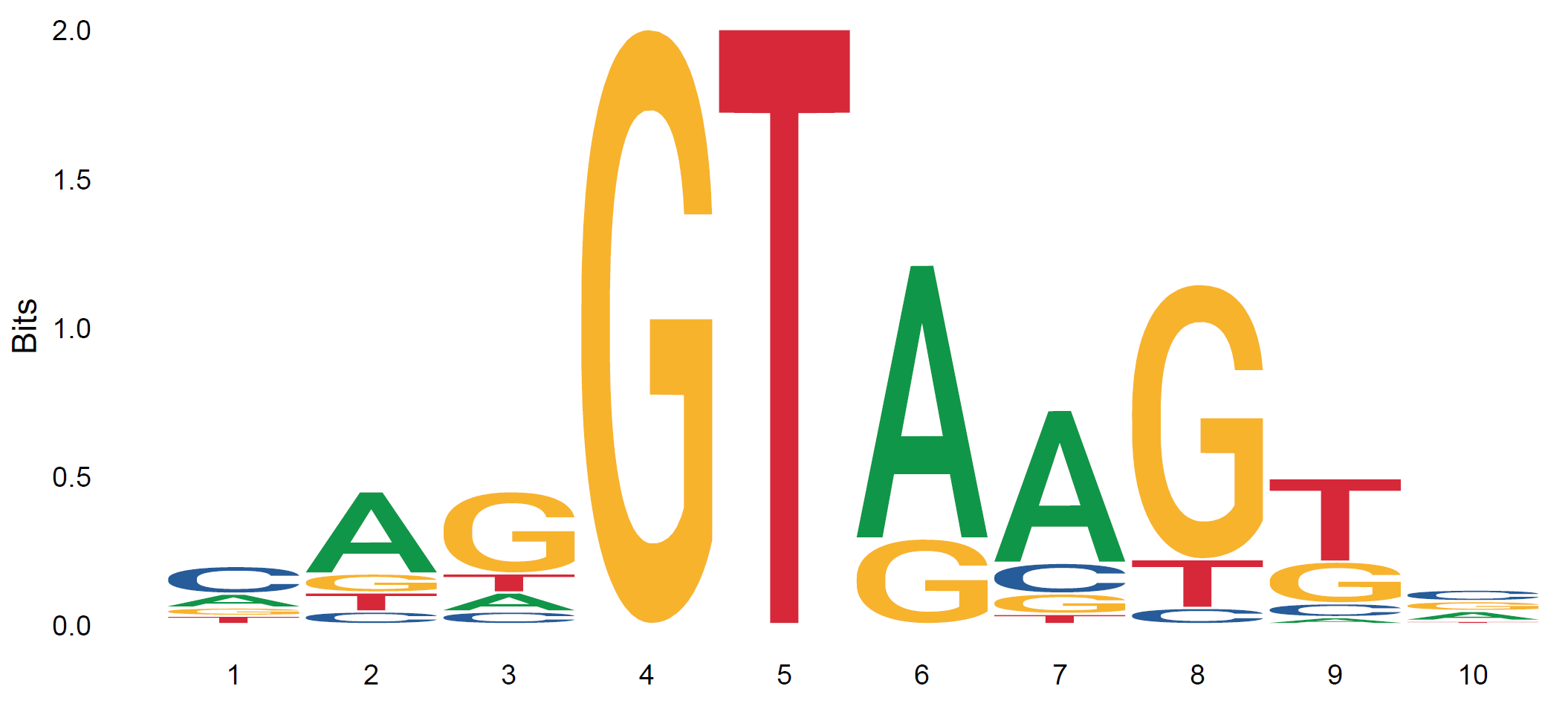
下载结果文件,解压后可得到7个文件,重点是PDF和PNG两种格式的图片。以“.os”为拓展名的文件是上传的原始序列,而“seqlogo.pfsm.matrix.xls”为position frequency matrix文件,其他为任务状态文件,可忽略。以下是两种常见的LOGO图。
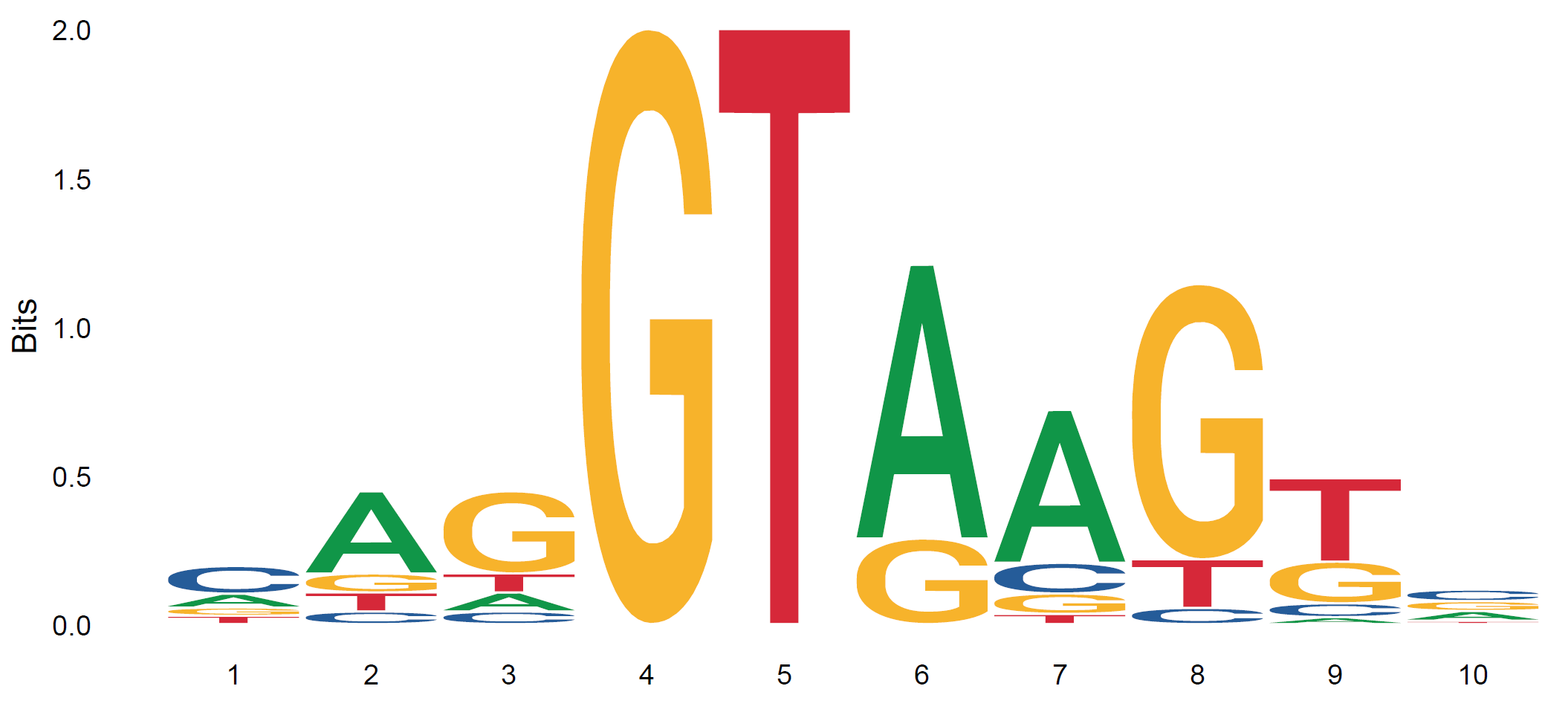
核酸序列LOGO图:单位选择为bits,配色方案选择默认,LOGO范围设置成3-12。
蛋白序列LOGO图:单位选择为Probability,配色方案选择自定义。

序列logo图由Tom Schneider 和Mike Stephens发明,用来分析和展示同源序列motif的保守性。图中的每个字母的高度与该位置的相应碱基或氨基酸残基的出现频率成正比,常以bits为单位。每个位置的字母按照保守性从大到小排列,可以方便的从顶端的字母识别保守序列,例如下图CAP的保守序列是“AA-TGTGA------ TCACA-TT”。

(Genome research, 2004;Bioinformatics, 2020)
参考文献
Tareen A, Kinney J B. Logomaker: beautiful sequence logos in Python[J]. Bioinformatics, 2020, 36(7): 2272-2274.
Crooks G E, Hon G, Chandonia J M, et al. WebLogo: a sequence logo generator[J]. Genome research, 2004, 14(6): 1188-1190.
下载示例文件,以示例文件1为例,点击选择文件按钮,上传范例序列,将LOGO范围设为3-12,其他参数保持默认,然后提交任务。
任务提交后,在左侧导航栏中点击我的项目查看任务进度,任务完成后点击下载按钮可下载绘图结果文件。
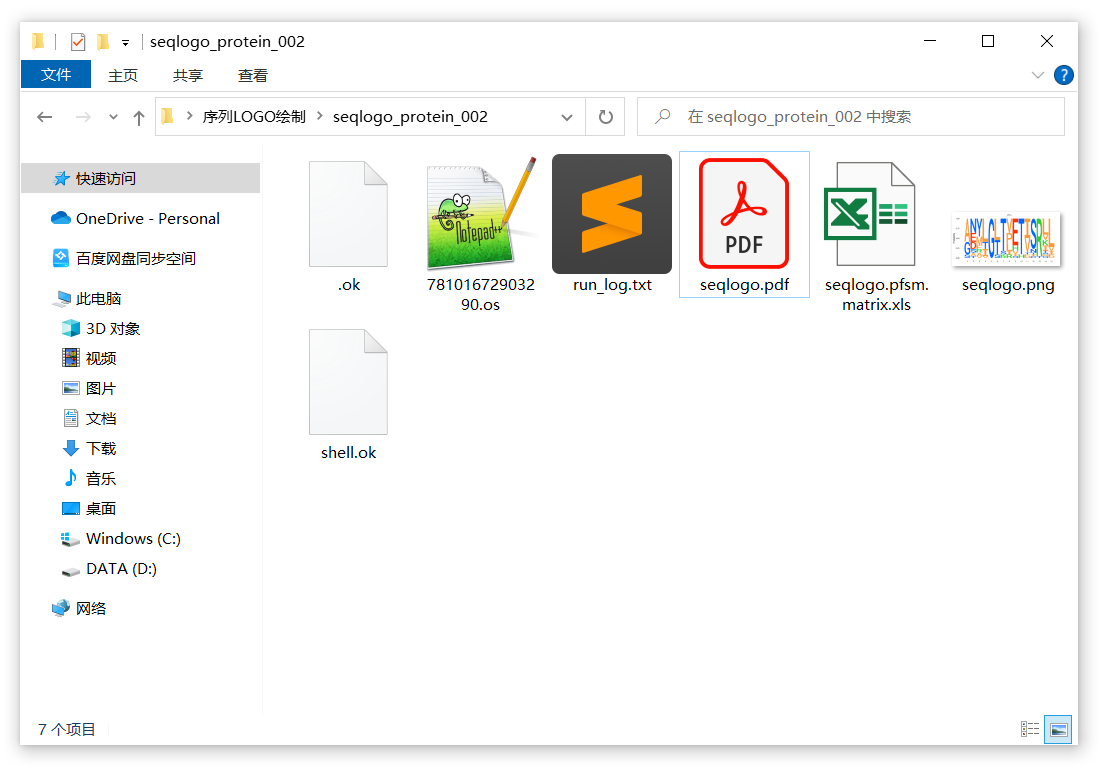
结果文件夹中主要包含7个文件,最重要的是PDF和PNG格式的图片,以“.os”为拓展名的文件为大家上传的原始数据,其他文件为任务状态文件。
默认绘制效果如下:
当然,我们也可以选择自定义配色方案,我们可以指定任意以#号开头的16进制色号,如下,这里的LOGO范围设为2-12。

绘制效果如下,颜色清新多了!

除了核酸序列,也可以使用序列LOGO工具展示蛋白序列,以示例文件2为例,点击选择文件按钮,上传范例序列,为了演示,这里将单位设为probability,配色方案选择自定义,我们也可以对氨基酸进行分组并指定颜色,其他参数保持默认,然后提交任务。
绘制效果如下:

至于对位序列文件的准备,其实,用于序列对位(multiple sequence alignment)的工具有很多,比如我喜欢使用MEGA中的Alinment功能,方法可选内置的Align by ClustalW或Align by MUSCLE。“对齐”后,将首尾两端“裁齐”。裁剪方法:框选不要的序列部分,按Delete键即可,然后将裁剪后的序列导出为fasta格式文件。

详细教程:
https://www.omicshare.com/forum/thread-4176-1-1.html
Q1. 上传的数据需要保存成什么格式?文件名称和拓展名有没有要求?
OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。如果是核酸、蛋白序列文件,必须为FASTA格式(本质是文本文件)。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”“.fasta”等,例如 mydata01.txt,gene02.xlsx 。
Q2. 提交时报错常见问题:
1.提交时显示X行X列空行/无数据,请先自查表格中是否存在空格或空行,需要删掉。
2.提交时显示列数只有1列,但表格数据不止1列:列间需要用分隔符隔开,先行检查文件是否用了分隔符。
其它提示报错,请先自行根据提示修改;如果仍然无法提交,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q3. 提交的任务完成后却不出图该怎么办?
主要原因是上传的数据文件存在特殊符号所致。可参考以下建议逐一排查出错原因:
(1)数据中含中文字符,把中文改成英文;
(2) 数据中含特殊符号,例如 %、NA、+、-、()、空格、科学计数、罗马字母等,去掉特殊符号,将空值用数字“0”替换;
(3)检查数据中是否有空列、空行、重复的行、重复的列,特别是行名(一般为gene id)、列名(一般为样本名)出现重复值,如果有删掉。
排查完之后,重新上传数据、提交任务。如果仍然不出图,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q4.下载的结果文件用什么软件打开?
OmicShare云平台的结果文件(例如,下图为KEGG富集分析的结果文件)包括两种类型:图片文件和文本文件。
图片文件:
为了便于用户对图片进行后期编辑,OmicShare同时提供位图(png)和矢量图(pdf、svg)两种类型的图片。对于矢量图,最常见的是pdf和svg格式,常用Ai(Adobe illustrator)等进行编辑。其中,svg格式的图片可用网页浏览器打开,也可直接在word、ppt中使用。
文本文件:
文本文件的拓展名主要有4种类型:“.os”、“.xls”、“.log”和“.txt”。这些文件本质上都是制表符分隔的文本文件,使用记事本、Notepad++、EditPlus、Excel等文本编辑器直接打开即可。结果文件中,拓展名为“.os”文件为上传的原始数据;“.xls”文件一般为分析生成的数据表格;“.log”文件为任务运行日志文件,便于检查任务出错原因。
Q5. 提交的任务一直在排队怎么办?
提交任务后都需要排队,1分钟后,点击“任务状态刷新”按钮即可。除了可能需运行数天的注释工具,一般工具数十秒即可出结果,如果超出30分钟仍无结果,请联系OS客服,发送任务编号给OmicShare客服,会有专人为你处理任务问题。
Q6. 结果页面窗口有问题,图表加载不出来怎么办?
尝试用谷歌浏览器登录OmicShare查看结果文件,部分浏览器可能不兼容。





 扫码支付更轻松
扫码支付更轻松



