PHI-base(Pathogen Host Interactions),病原宿主互作数据库,是一个免费开放的数据库,收录了经过实验验证或文献报道的能够感染植物、动物、真菌和昆虫的真菌、卵菌、细菌等病原菌的致病基因、毒力基因和效应蛋白基因。另外,数据库还收录了抗真菌化合物及其靶基因。
截至2018.11,最新版本为4.6,共收录6438个基因、11340对互作关系、263个病原菌、194个宿主、510种疾病。使用DIAMOND默认参数进行比对。
输入文件
上传fasta格式的核酸或蛋白query序列文件。
结果
1.序列注释结果总表
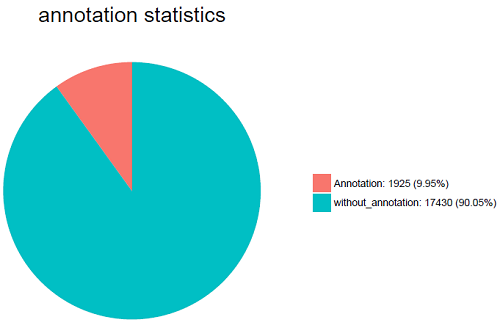
2.比对结果统计饼图
1)将比对上的与比对不上的序列数目进行统计并画饼图。
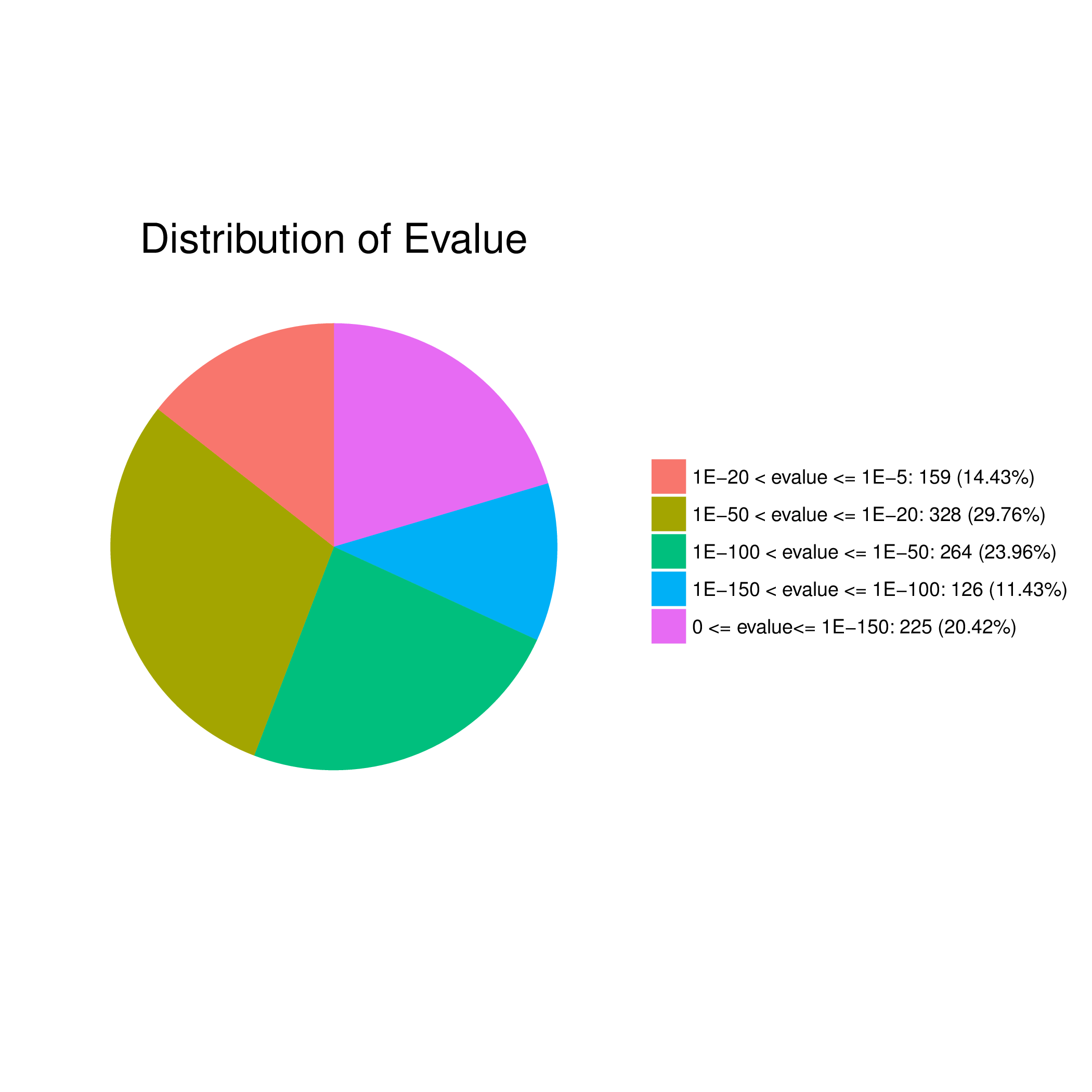
2)E-value分布图
E值是指期望数据库中具有某一统计学意义配对序列的值,可理解为比对的假阳性率。E值越小,结果越可靠。对E值分为5个范围进行统计,并画饼图。
1. 比对结果总表
表头解释:
Query_id:查询序列编号
Subject_id:PHI数据库对应基因编号
Query_start:查询序列比对起始位点
Query_end:查询序列比对终止位点
Subject_start:比对上的部分在在数据库序列上的起始位点
Subject_end:比对上的部分在在数据库序列上的终止位点
Align_length:比对上的长度
Positive:比对上的氨基酸或碱基数
Gap:Gap 的数目
Coverage: query比对上subject序列的覆盖度
Identity(%):序列相似度
E_value:比对E值,越低表示可靠性越高
Score:比对的打分(打分越高,可信度越高)
DB_Type:参考基因的数据库来源
Accession:来源数据库对应编号
Gene_name:基因名称
Pathogen_NCBI _ID:病原菌NCBI物种分类ID
Pathogen_species:病原菌物种
Disease_name:所引起疾病
Host_Descripton:宿主描述信息
Host_NCBI_ ID:宿主NCBI物种分类ID
Experimental_host:宿主名称
Function:蛋白功能
2. 比对结果统计饼图
将比对上的与比对不上的序列数目进行统计并画饼图。
3. E值分布统计
E值是指期望数据库中具有某一统计学意义配对序列的值,可理解为比对的假阳性率。E值越小,结果越可靠。对E值分为5个范围进行统计,并画饼图。
Q1. 上传的数据需要保存成什么格式?文件名称和拓展名有没有要求?
OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。如果是核酸、蛋白序列文件,必须为FASTA格式(本质是文本文件)。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”“.fasta”等,例如 mydata01.txt,gene02.xlsx 。
Q2.注释工具的任务一般要跑多久?
注释工具的任务时长一般与提交的序列条数成正比,且不同工具耗时也差异很大。例如GO功能注释近一年来所有任务平均时长约为2天,而NR注释工具近一年来所有任务平均时长约为3小时。一般情况下,如果任务耗时超出一周可联系OS客服,发送任务编号给OmicShare客服,会有专人为你处理任务问题。





 扫码支付更轻松
扫码支付更轻松


