功能说明
Gene Ontology(简称GO)是一个国际标准化的基因功能分类体系,提供了一套动态更新的标准词汇表(controlled vocabulary)来全面描述生物体中基因和基因产物的属性。GO总共有三个ontology(本体),分别描述基因的分子功能(molecular function)、细胞组分(cellular component)、参与的生物过程(biological process)。GO的基本单位是term(词条、节点),每个term都对应一个属性。
输入文件
1.选择文件
输入fasta格式的核酸或蛋白query序列或上传fasta格式的query序列文件。
每个序列前需带有“>序列名”,且序列名不能带有特殊字符及空格;序列间需要回车“Enter”间隔一行,不能带有空格。
示例:
2. 选择物种
选择GO数据库的数据集。
注意:注释工具视序列数需时较长,GO注释如果选择数据库为“所有物种”,时间会加长。请耐心等待任务完成~如果任务显示出错,可以联系平台OmicShare客服处理。
输出结果
(具体图例、统计表包含信息请见“例子”栏)
1 注释结果
将注释上的与注释不上的序列数目进行统计并画饼图。
表1 注释结果数目统计表
图1 注释结果数目统计饼图
2. GO功能分类统计
表2 GO功能分类信息统计表
图2 GO 功能分类统计图
输出结果说明
表1 注释结果数目统计表表头解释
Total sequence number:输入的所有序列数
Annotation sequences:注释上的序列数
Without annotation sequences:没有注释上的序列数
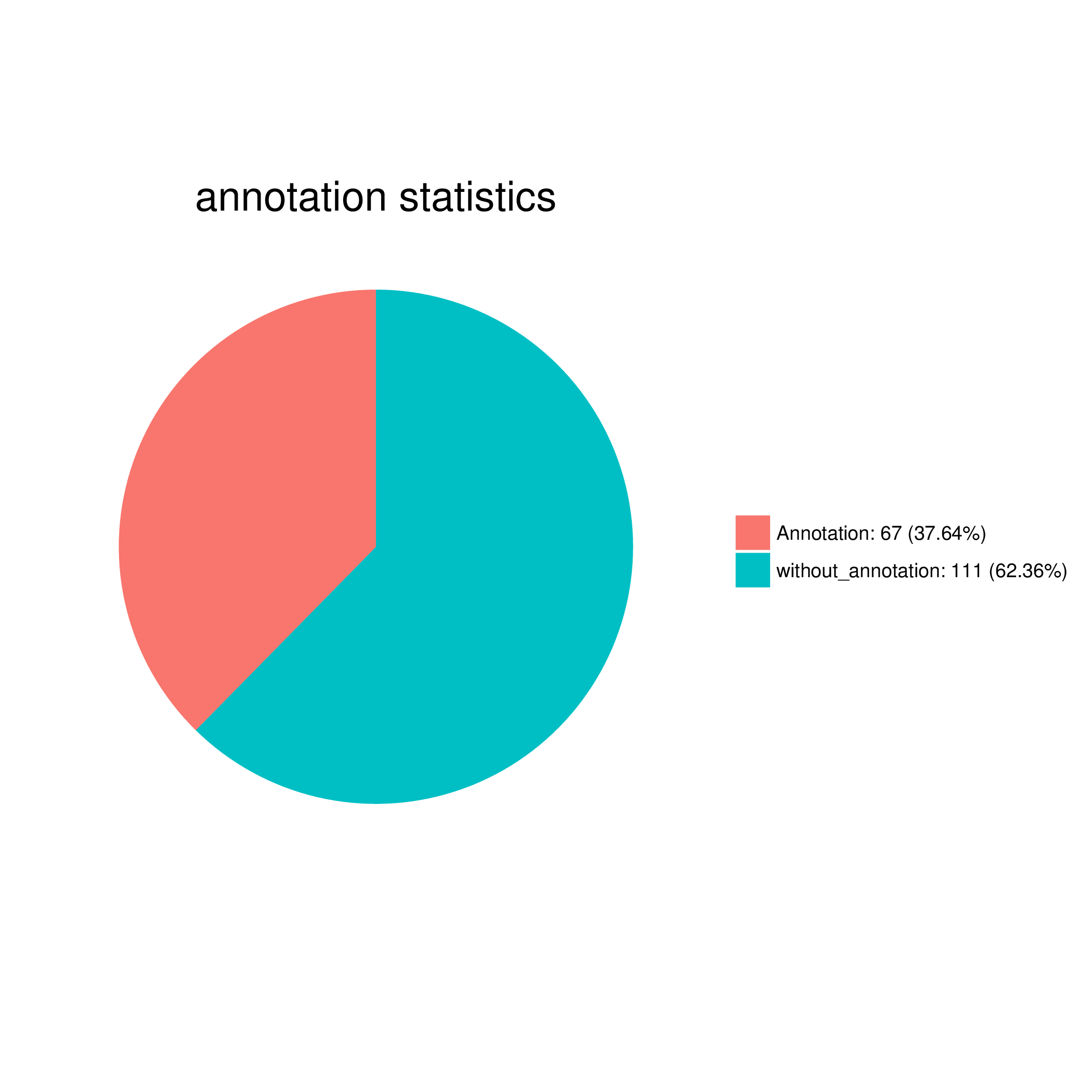
图1 注释结果数目统计饼图
表2 GO功能分类信息统计表表头解释
GO ID (level1):GO本体的类别ID (biological_process 或 cellular_component 或 molecular_function)
GO Term (level1):GO本体的类别 (biological_process 或 cellular_component 或 molecular_function)
GO ID (level2):GO条目ID
GO Term (level2):GO条目
number_of_all (All):Unigene在注释到各GO条目中的数量
Genes_of_all:注释到该GO条目的Gene ID
![]()
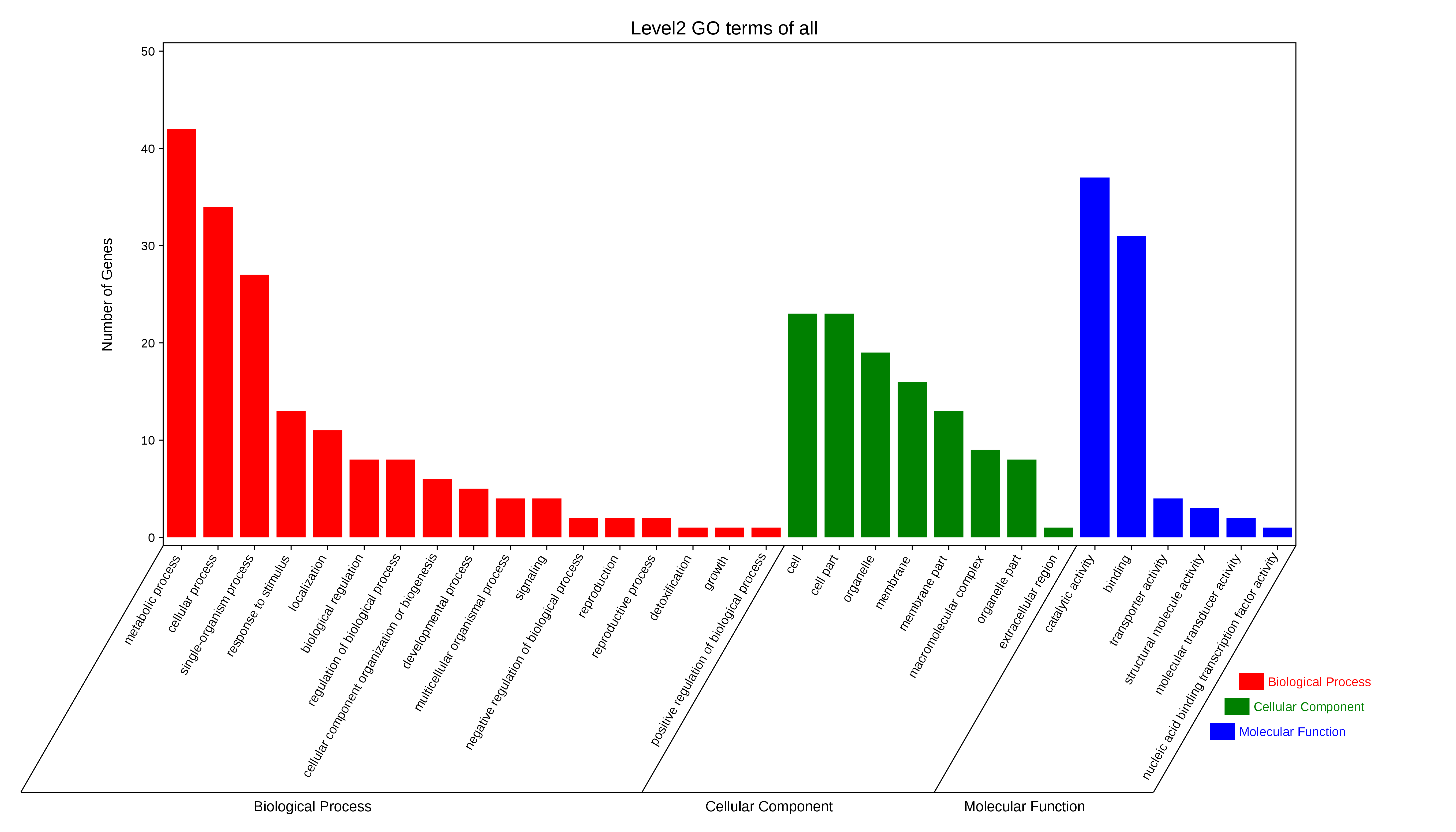
图2 GO 功能分类统计图
Q1. 上传的数据需要保存成什么格式?文件名称和拓展名有没有要求?
OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。如果是核酸、蛋白序列文件,必须为FASTA格式(本质是文本文件)。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”“.fasta”等,例如 mydata01.txt,gene02.xlsx 。
Q2.注释工具的任务一般要跑多久?
注释工具的任务时长一般与提交的序列条数成正比,且不同工具耗时也差异很大。例如GO功能注释近一年来所有任务平均时长约为2天,而NR注释工具近一年来所有任务平均时长约为3小时。一般情况下,如果任务耗时超出一周可联系OS客服,发送任务编号给OmicShare客服,会有专人为你处理任务问题。





 扫码支付更轻松
扫码支付更轻松



