功能:
利用CCA/RDA分析环境因子对群落分布影响。
输入:
1. 输入文件:
(1)OTU丰度表(必须)
(2)各样本的环境因子表格,要求环境因子数目小于样本数(必须)
(3)如果有分组信息,请输入样本分组信息表。单样本最多支持两种分组信息,如A样本同时属于病人分组,也属于成年人分组。(非必需)
(4) OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。
参数:
分析类型:
可以选择CCA分析 或者 RDA分析
是否显示物种名称:
默认显示样本名称
前几物种展示:
默认该分类水平下丰度在前10的微生物作图
图形名称:
设定图形标题名称,暂不支持中文
点的大小:
图形点大小控制,默认为3,值越大点越大
点透明度:
图形点透明度控制,默认为0.7,可以填0-1的值
自定义分组颜色(颜色的个数要和分组个数相等)
设定分组颜色,所选择颜色数目应与分组数目一致
输出:
1) 样本坐标表: 记录了样本在各个维度上的位置
2) 物种坐标表: 记录了物种在各个维度上的位置
3) 环境因子坐标: 记录了环境因子在各个维度上的位置
4) 环境因子相关性表: 前两列表示环境因子箭头与排序轴夹角余弦值,表示环境因素与排序轴相关性。
5) CCA/RDA结果图
CCA1-2.nonlabs.png/pdf: A CCA/RDA plot without sample names
CCA1-2.png/pdf: A CCA/RDA plot with sample names
案例演示:
输入: OTU丰度表 环境因子列表文件 分组信息表
输出:
1:样本坐标表
Samples CCA1 CCA2 CCA3 CCA4 CCA5 CA1
CK1 -1.17237086899951 0.231164561576716 2.43536547360125 0.468613538470372 -0.521936162667495 -0.808046986758666
CK2 -1.1572665823102 -1.42860877240687 -0.549032209802859 2.83351063677702 1.43378119851602 2.02534821080922
CK3 -1.49570686760746 0.0273462956558565 -0.706329227389229 0.865838710760632 -0.290912650451685 1.01877976615298
CK4 -1.20710868460347 0.370935253362443 -2.60560316720684 -2.42810618842451 0.168506841308157 -1.6719608268434
CK5 -1.5562702802342 0.651269590349488 0.595882020371293 -1.45146930145046 -0.318000696274572 -0.251630348306944
CA1 0.974610196394923 -0.104745619111151 -0.334203278424584 1.04120295623205 -0.0531562686390503 -0.942931875531439
CA2 0.760730059809399 -2.12637958035229 0.745781118766851 -1.63166065483332 -0.0165817224481634 -0.573209913163789
CA3 0.80435142753797 0.700368060885899 -0.263211126786403 0.229812203163464 -1.61999680223531 0.0204114357736938
CA4 0.567093187244471 0.262752025306537 0.0148235934121042 -0.437995730700719 -0.822034862380148 0.231909808702752
CA5 0.700914846513469 1.98976523732017 -0.162807163417265 0.0466015118241849 3.06827940812714 1.3262899501884
2:物种坐标表
Species CCA1 CCA2 CCA3 CCA4 CCA5 CA1
Alicyclobacillus 0.34595108156025 -0.249931443439814 -0.00508892535787803 0.0575514559218589 -0.0294336770660223 -0.168446818978494
Bacillus -0.434908707180272 0.110247143354924 0.143966641687111 -0.0483216220508095 -0.0280734921997281 -0.0313666148504983
CandidatusRhabdochlamydia 0.332412329234085 0.149552158756664 -0.110963677277933 -0.00513846687288351 -0.0755745296229585 0.0185259066401049
CandidatusSolibacter -0.338931730698679 0.0879930888338368 -0.0635812894580597 -0.220352060529118 -0.00527399612415062 -0.0720185430737823
Mycobacterium 0.30769780457627 0.109805580124376 0.0324946604566324 0.015709933373511 0.251544295518129 0.0327943021149191
Acidobacterium 0.593748839228754 0.413222114904331 -0.0190275868032072 0.0499162197773069 0.154477266462497 0.146438254658603
CandidatusKoribacter -0.807533540490883 -0.238088566626537 -0.166963852695348 0.255945631985289 0.07726025494122 0.691926396129785
Conexibacter 0.682330827072613 0.380118852507714 0.0317777784942295 -0.0492734196140707 0.000966010984764718 0.205043073765086
Nocardioides -0.820114462482764 0.0737283559695164 0.673717900416305 0.279984911111747 0.0122300186916335 0.036916331507856
Pilimelia -1.27268641146115 -0.0237692247359973 -0.375484360703181 -0.0268117477942791 0.0397218484000157 -0.0137762143280586
Sinomonas -1.1647948301215 -0.0991385843491781 -0.140260843226405 0.265065165234655 -0.058581660637485 0.625884640720445
Streptomyces -0.706743539907205 -0.0165173955314497 -0.210856929356171 -0.0941302900650263 0.027518630271319 0.102114406891466
Flavobacterium 0.197779672222041 -1.21998311475552 0.276998901549968 -0.4668873108256 0.147784962249983 0.0874168205447417
Kitasatospora -0.513045251422147 -0.426617698321745 -0.149095388924391 0.188923417085328 0.176398426958694 0.490444761685818
Parachlamydia 0.490302339839949 0.0763178809781672 -0.126739126101196 -0.0644678365797921 -0.240110777182143 0.0305911858454124
Rathayibacter 0.543807338526059 0.155078631329423 0.19654117415888 0.0411930207557103 0.151295881530674 -0.0191993792585311
Edaphobacter 0.499205261603634 0.353178206304564 0.0594334945395013 -0.116917048551099 0.283954689966612 0.41768442620536
Catellatospora -1.41823522496713 -0.127184907485305 -1.38322918921647 -0.398764248425288 0.174072239090334 -0.655892489276133
Virgisporangium -1.3141321649437 -0.0283163960812968 -1.07500687863742 -0.394924480802182 0.13068384248966 -0.783230745347689
Prevotella 0.719998724070353 -1.50861319724635 0.529239772332071 -1.03135529651776 0.213618328885839 -0.564226755041147
3:环境因子坐标
Constrain CCA1 CCA2 CCA3 CCA4 CCA5 CA1
AN 0.215918459134623 0.300454636633771 0.0037843269271663 0.189483055402639 -0.909498807539178 0
AP -0.88321103447841 -0.00780260484008147 0.179211133562975 0.393625808498019 0.18116147610527 0
AK -0.948073637913666 0.0805585577858008 0.294316730344931 0.066086075520977 0.0606381787484189 0
OM -0.256087308224969 0.643580255179155 0.0716367791250762 0.502223686811577 0.51270194654555 0
Ph -0.945408617807588 -0.0121426079324861 -0.30469138398077 -0.0743625293392954 0.0876839620791046 0
4:DCA结果表
DCA Eigenvalues Decorana_values Axis_lengths
DCA1 0.235586191937275 0.24189609354078 2.04870842887944
DCA2 0.080053434386399 0.046972558162257 2.25698204351043
DCA3 0.0563583935717481 0.0158306521768173 1.47759325956464
DCA4 0.0261967709460555 0.00724315524884521 1.65694736271154
5:环境因子相关性表
Constrain RDA1_cos RDA2_cos
AN 0.583576682528919 0.812058037093742
AP -0.999960979319677 -0.00883401596283277
AK -0.996409414613128 0.0846656864988553
OM -0.369716370113742 0.929144663478147
Ph -0.999917529028949 -0.0128427076833458
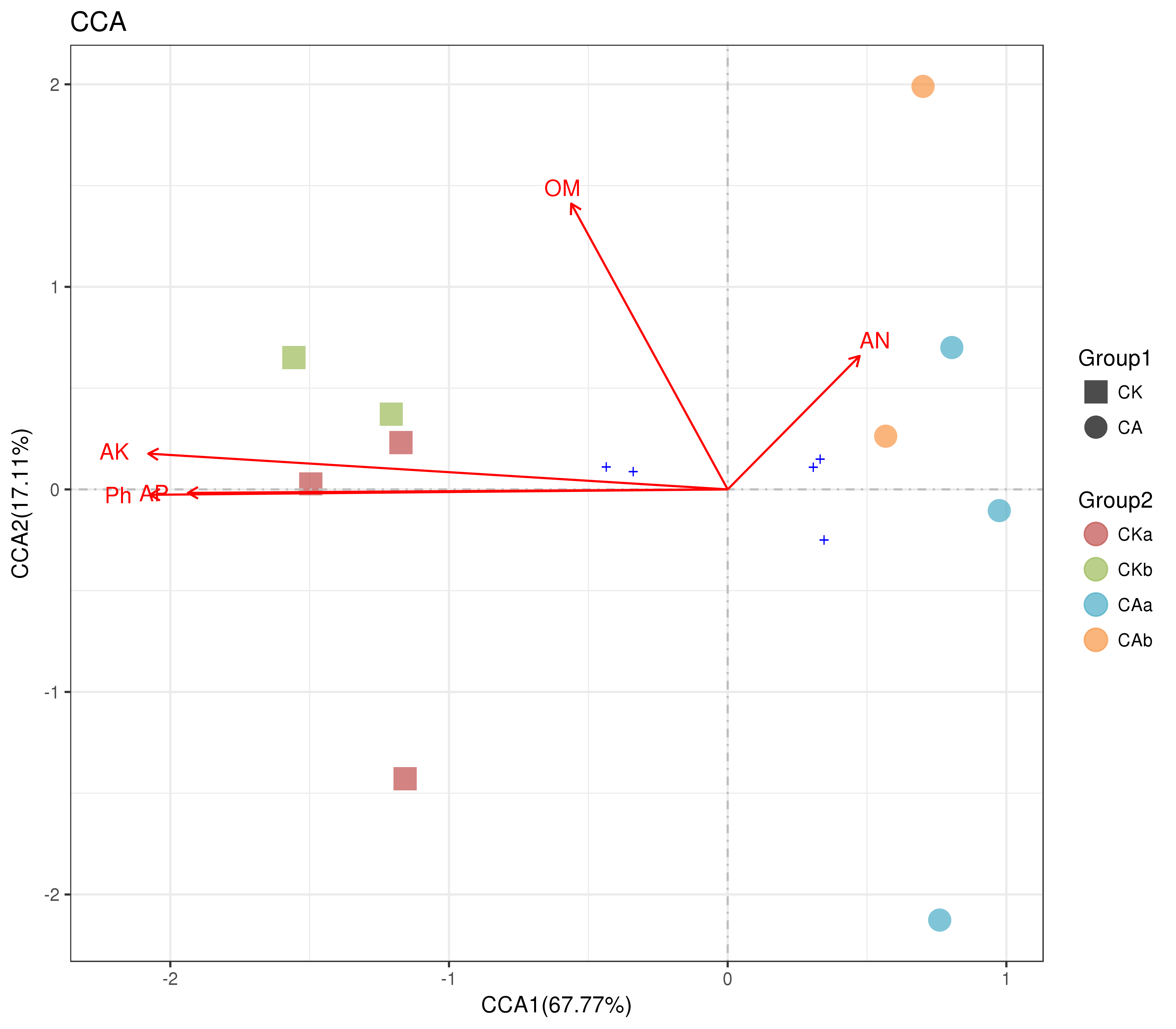
CCA/RDA结果图:
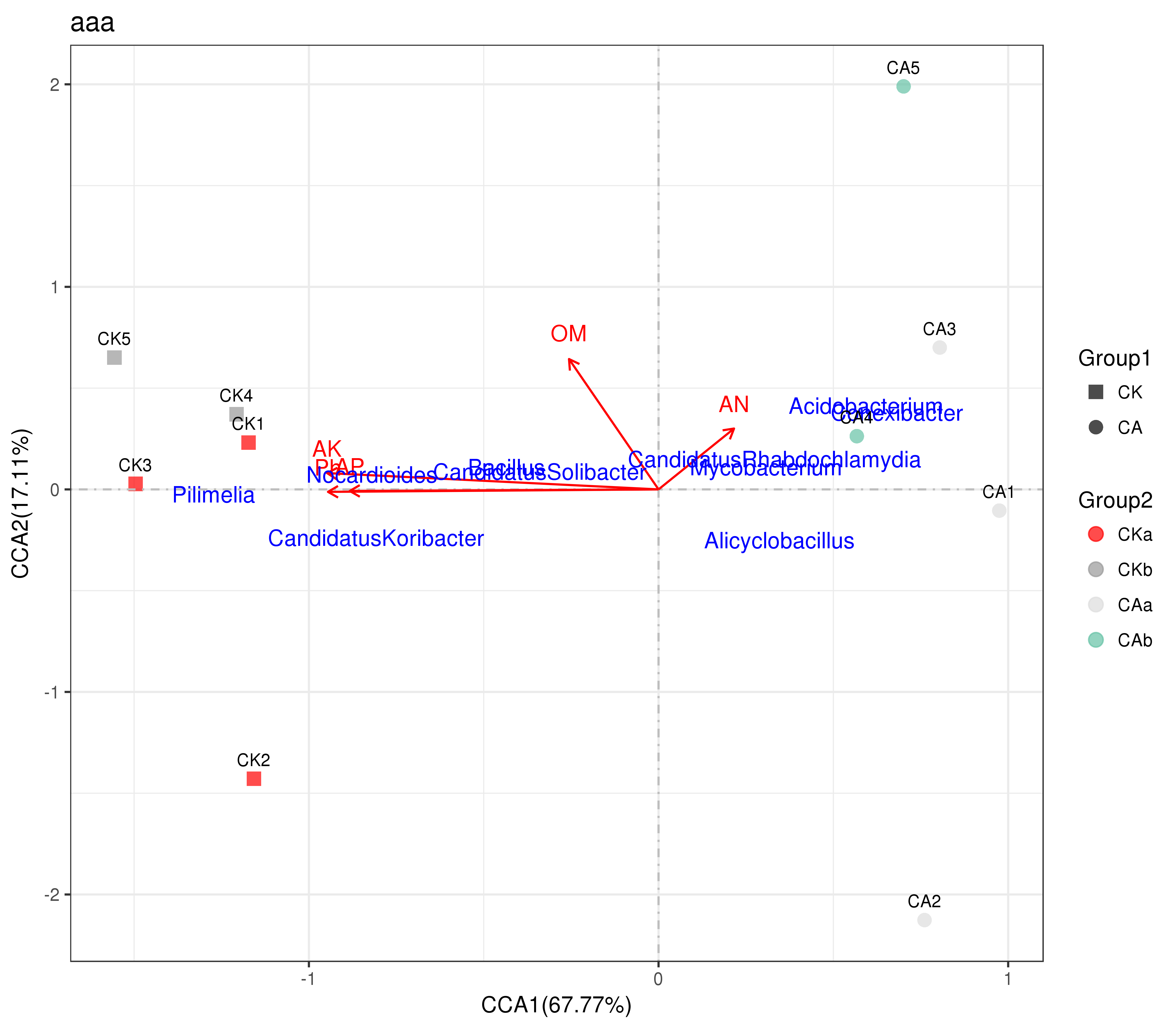
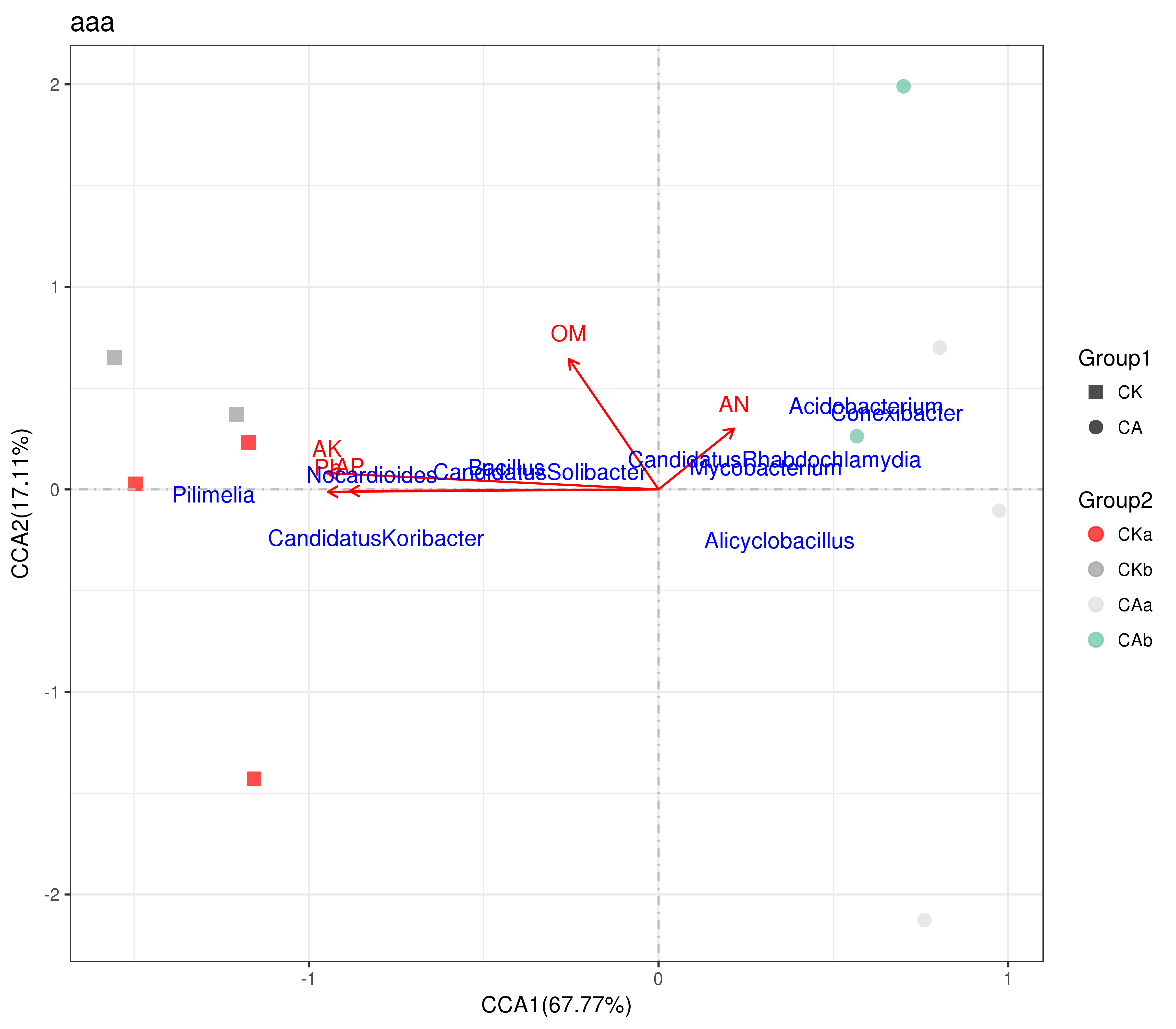 。
。
Q1. 上传的数据需要保存成什么格式?文件名称和拓展名有没有要求?
OmicShare当前支持txt(制表符分隔)文本文件、csv(逗号分隔)文本文件、以及Excel专用的xlsx格式,同样支持旧版Excel的xls(Excel 97-2003 )格式。如果是核酸、蛋白序列文件,必须为FASTA格式(本质是文本文件)。
文件名可由英文和数字构成,文件拓展名没有限制,可以是“.txt”、“.xlsx”、“.xls”、“.csv”“.fasta”等,例如 mydata01.txt,gene02.xlsx 。
Q2. 提交时报错常见问题:
1.提交时显示X行X列空行/无数据,请先自查表格中是否存在空格或空行,需要删掉。
2.提交时显示列数只有1列,但表格数据不止1列:列间需要用分隔符隔开,先行检查文件是否用了分隔符。
其它提示报错,请先自行根据提示修改;如果仍然无法提交,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q3. 提交的任务完成后却不出图该怎么办?
主要原因是上传的数据文件存在特殊符号所致。可参考以下建议逐一排查出错原因:
(1)数据中含中文字符,把中文改成英文;
(2) 数据中含特殊符号,例如 %、NA、+、-、()、空格、科学计数、罗马字母等,去掉特殊符号,将空值用数字“0”替换;
(3)检查数据中是否有空列、空行、重复的行、重复的列,特别是行名(一般为gene id)、列名(一般为样本名)出现重复值,如果有删掉。
排查完之后,重新上传数据、提交任务。如果仍然不出图,可通过左侧导航栏的“联系客服”选项咨询OmicShare客服。
Q4.下载的结果文件用什么软件打开?
OmicShare云平台的结果文件(例如,下图为KEGG富集分析的结果文件)包括两种类型:图片文件和文本文件。
图片文件:
为了便于用户对图片进行后期编辑,OmicShare同时提供位图(png)和矢量图(pdf、svg)两种类型的图片。对于矢量图,最常见的是pdf和svg格式,常用Ai(Adobe illustrator)等进行编辑。其中,svg格式的图片可用网页浏览器打开,也可直接在word、ppt中使用。
文本文件:
文本文件的拓展名主要有4种类型:“.os”、“.xls”、“.log”和“.txt”。这些文件本质上都是制表符分隔的文本文件,使用记事本、Notepad++、EditPlus、Excel等文本编辑器直接打开即可。结果文件中,拓展名为“.os”文件为上传的原始数据;“.xls”文件一般为分析生成的数据表格;“.log”文件为任务运行日志文件,便于检查任务出错原因。
Q5. 提交的任务一直在排队怎么办?
提交任务后都需要排队,1分钟后,点击“任务状态刷新”按钮即可。除了可能需运行数天的注释工具,一般工具数十秒即可出结果,如果超出30分钟仍无结果,请联系OS客服,发送任务编号给OmicShare客服,会有专人为你处理任务问题。
Q6. 结果页面窗口有问题,图表加载不出来怎么办?
尝试用谷歌浏览器登录OmicShare查看结果文件,部分浏览器可能不兼容。
引用OmicShare Tools的参考文献为:
Mu, Hongyan, Jianzhou Chen, Wenjie Huang, Gui Huang, Meiying Deng, Shimiao Hong, Peng Ai, Chuan Gao, and Huangkai Zhou. 2024. “OmicShare tools: a Zero‐Code Interactive Online Platform for Biological Data Analysis and Visualization.” iMeta e228. https://doi.org/10.1002/imt2.228案例1:
发表期刊:Environmental Science & Technology
影响因子:11.4
发表时间:2023
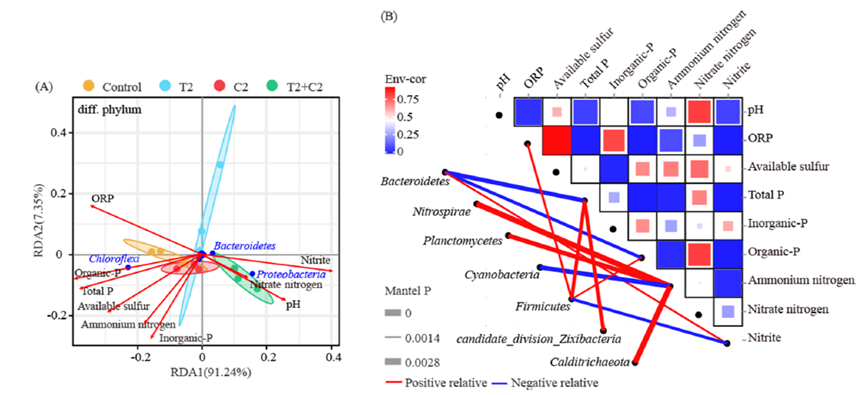
Figure 4. RDA/CCA (A) and network heatmap (B) analysis of the relationship between microbial abundance and environmental factors (phylum level, top 15; Pearson correlation coefficient > 0.8 or < −0.8, P < 0.05).
引用方式:The OmicShare platform was used for clustering heatmaps, NMDS, and RDA analysis (https://www.omicshare.com/tools/).
参考文献:
Lai J, Li Z, Wang Y, et al. Tritium and Carbon-14 Contamination Reshaping the Microbial Community Structure, Metabolic Network, and Element Cycle in the Seawater Environment[J]. Environmental Science & Technology, 2023, 57(13): 5305-5316.
案例2(CCA)
发表期刊:Journal of Hazardous Materials
影响因子:13.6
发表时间:2022
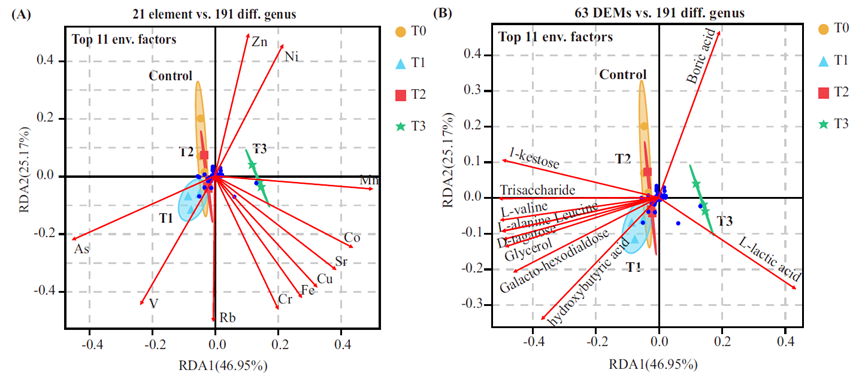
Fig. 10. RDA/CCA analysis (A, B)
引用方式:
The RDA/CCA analysis was performed using Omicshare online tools(www.omicshare.com/tools).
参考文献:
Fu Q, Lai J, Ji X, et al. Alterations of the rhizosphere soil microbial community composition and metabolite profiles of Zea mays by polyethylene-particles of different molecular weights[J]. Journal of Hazardous Materials, 2022, 423: 127062.
案例3(CCA)
发表期刊:Food & Function
影响因子:6.1
发表时间:2023
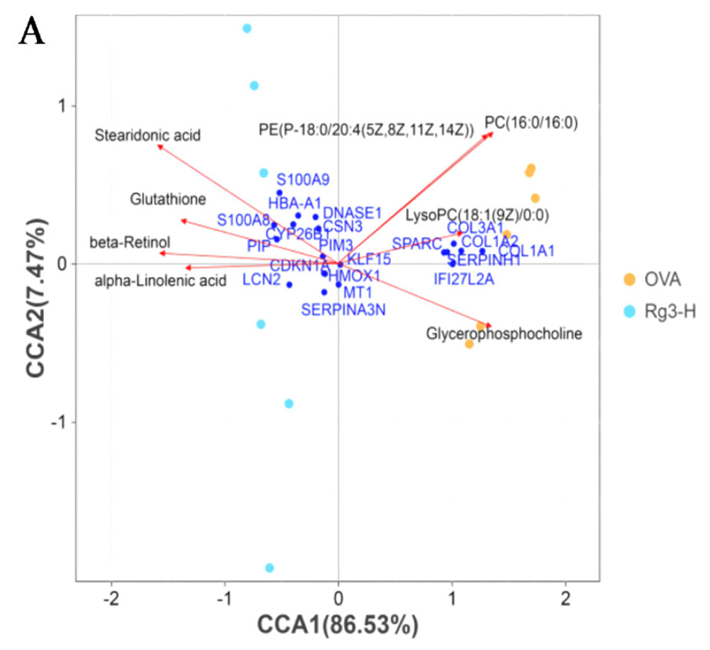
Fig. 4 (A) CCA of top 20 DEGs and 8 differential metabolites in AR mice treated with Rg3.
引用方式:
CCA was performed to mine the correlation between sets of variables of multiomics
data.32 The correlation of DEGs and differential metabolites was measured by using CCA. The content levels of differential metabolites and the expression levels of DEGs were imported
into software Canoco 5.0 (https://www.canoco5.com/) and the OmicShare database (https://omicshare.com/) with default parameters for CCA.
参考文献:
Liu J, Yang N, Yi X, et al. Integration of transcriptomics and metabolomics to reveal the effect of ginsenoside Rg3 on allergic rhinitis in mice[J]. Food & Function, 2023, 14(5): 2416-2431.





 扫码支付更轻松
扫码支付更轻松