功能:
计算表格内所有元素的两两相关性。
输入:
(1)表格包含分析数据及表头信息,如示例文件中的基因表达量表格,表头为样本名称,列名为基因ID,每行数据为对应基因在各样本中表达量的FPKM值
(2)输入文件为tab-delimited所保存的txt格式
参数:
(1)分析类型:包含两种,pearson和spearman,1)pearson系数,通常用于计算连续数据的相关性;2)spearman系数,专门用于计算等级数据的相关性。
(2)计算向量:默认列向量col。如示例文件中,若选择列向量,则以列为基础,计算每两列数据的相关性,即样本之间的相关性,可用于评估样本分组差异、样本重复性等;若选择行向量,则以行为基础,计算每两行数据的相关性,即基因之间的相关性,可用于评估基因的关联特征等。
(3)颜色,可根据数据量大小、相关性系数波动范围等选择2种或3种颜色,默认2种, white,green。
(4)字体大小:默认为10
(5)在格子上显示数字:默认为no,不显示
(6)画出格子边界:默认为no,不显示
(7)结果是否显示行名:默认为yes,显示
(8)结果是否显示列名:默认为yes,显示
输出:
all.cor.matrix.xls:相关系数矩阵,绘制热图的数据格式
all.pvalue.matrix.xls:相关显著性p值矩阵
all.cor_pvalue.list.xls:相关系数及p值列表,云平台工具绘制网络图的数据格式(网络图链接 )
all.cor_heatmap.png/pdf: 默认输出两种格式的结果图
结果说明:
相关系数为正表示正相关,为负表示负相关。常用的相关性强弱判断标准如下图:
相关系数绝范围 相关性强弱程度 0.8-1.0 极强相关 0.6-0.8 强相关 0.4-0.6 中等程度相关 0.2-0.4 弱相关 0.0-0.2 极弱相关或无相关
相关显著性p <0.05,代表显著相关;p <0.01,代表极显著相关
案例演示:
1、输入文件cor.txt2、参数
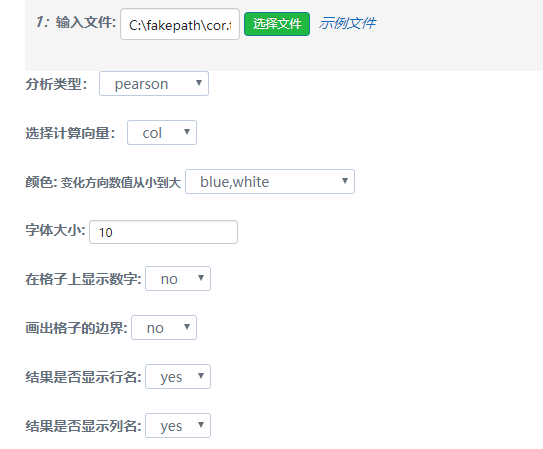
输出结果:
all.pearson.xls: all.pearson.xls
all.corMatrix.xls: all.corMatrix.xls
all.pMatrix.xls: all.pMatrix.xls
all.cor_heatmap.png/pdf
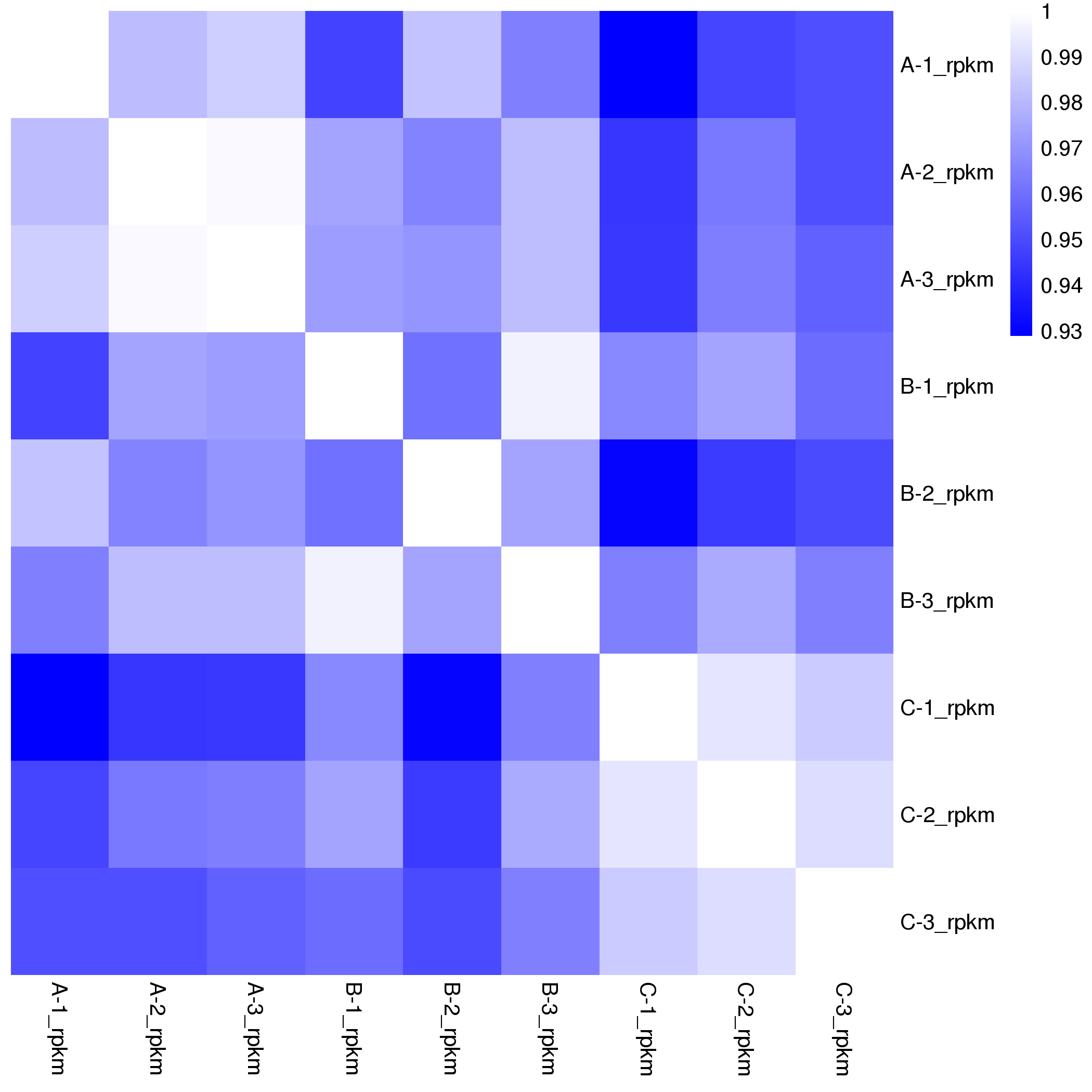




 扫码支付更轻松
扫码支付更轻松


