宏基因组测序,无目标片段的扩增步骤,直接基于样本总DNA进行测序分析的组学方法,因而分析对象是群落内所有微生物的全部DNA片段。可同时开展群落更精细的物种(如,种水平)和功能研究,除了经典的微生物群落分析方法,基于binning,CAG等高级分析,还可进行不依赖分离培养的“单菌”研究。
已广泛应用于人、动植物共生微生物,环境微生物等更精细的物种分析和功能分析,对于微生物分子机制的深入探讨有更强的应用价值。
该系列课程围绕宏基因组测序相关的技术特色、软件原理、研究思路、文献应用等方面开展分享交流。希望为大家提升宏基因组研究深度提供借鉴参考。
宏基因组测序,无目标片段的扩增步骤,直接基于样本总DNA进行测序分析的组学方法,因而分析对象是群落内所有微生物的全部DNA片段。可同时开展群落更精细的物种(如,种水平)和功能研究,除了经典的微生物群落分析方法,基于binning,CAG等高级分析,还可进行不依赖分离培养的“单菌”研究。
已广泛应用于人、动植物共生微生物,环境微生物等更精细的物种分析和功能分析,对于微生物分子机制的深入探讨有更强的应用价值。
本系列课程围绕宏基因组测序相关的技术特色、软件原理、研究思路、文献应用等方面开展分享交流。课程将根据需要更新,目前主要内容如下:
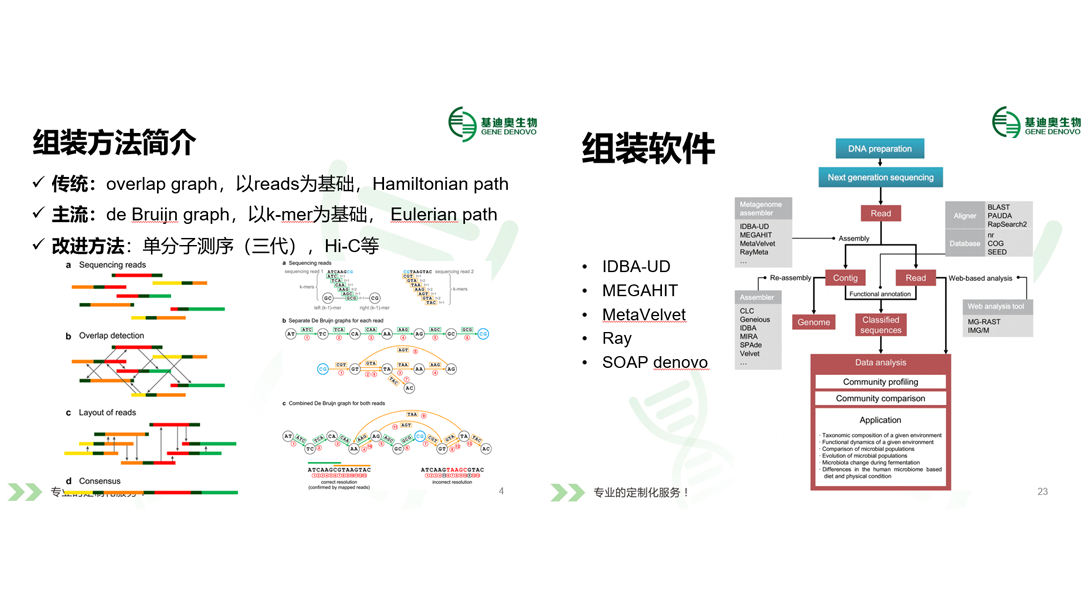
宏基因组组装原理讨论课程主要介绍宏基因组组装的主流方法原理、问题及选择方案,宏基因组组装难题和解决,以及介绍宏基因组组装软件;
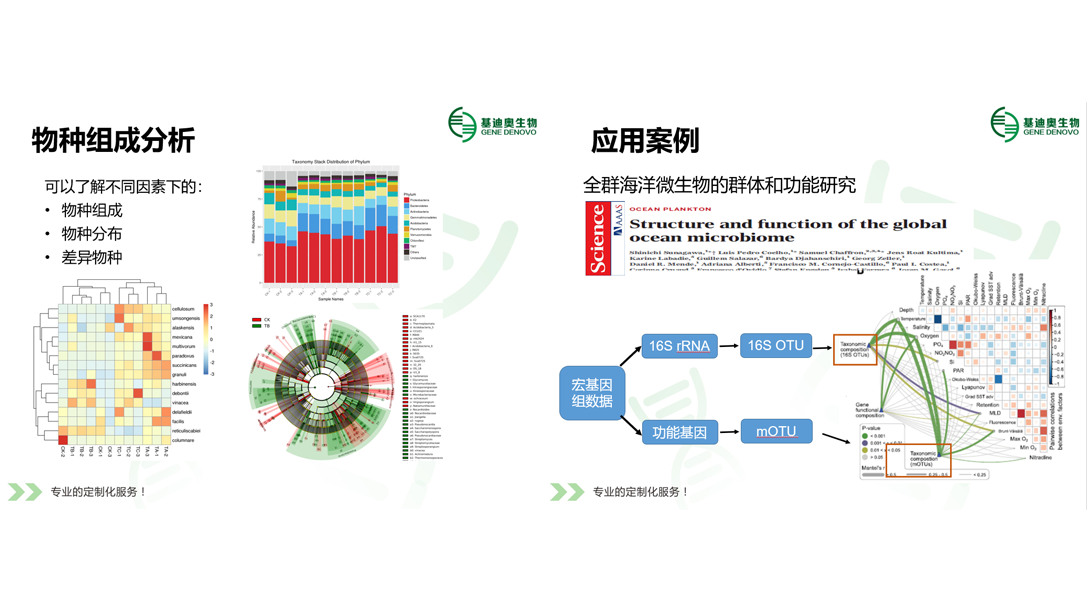
宏基因组物种注释方法介绍课程主要介绍基于特殊基因片段注释(结合案例对比宏基因组与16S测序)和基于广泛基因组序列注释(基于Reads比较软件比对、基于kmer检索软件比较);
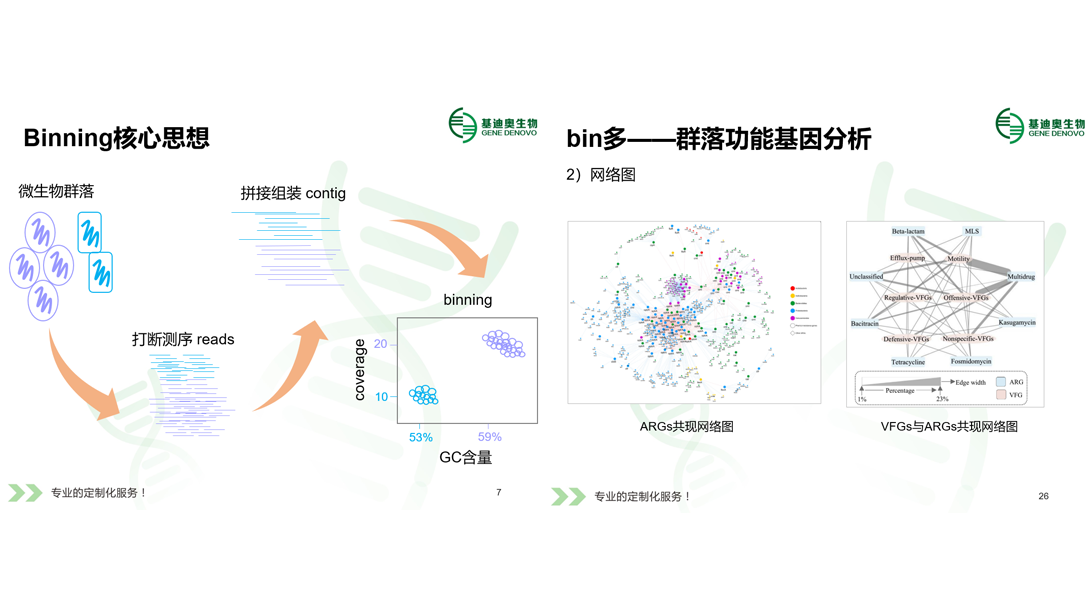
宏基因组binning软件原理和应用课程主要对比介绍binning软件Concoct、MaxBin2、MetaBAT2的原理及使用,同时结合案例介绍binning的研究思路和应用;
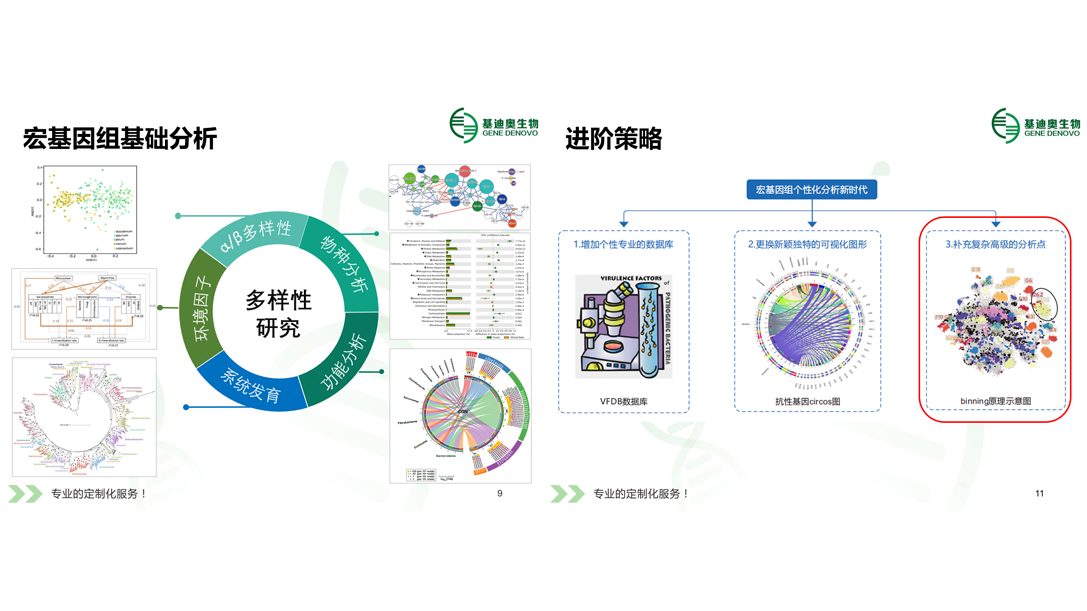
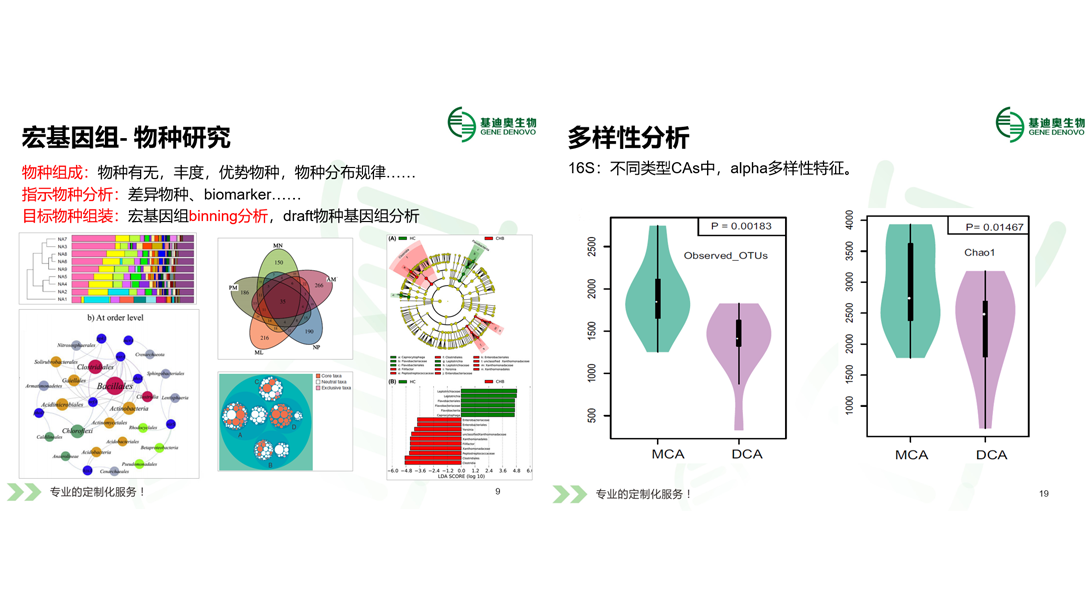
宏基因组数据库和图形升级优化策略课程从宏基因组的主要背景知识:宏基因组多样性研究、物种研究、功能研究、系统发育树研究、环境因子关联分析等,结合经典案例介绍宏基因组分析方法和相关图形;
宏基因组高级分析binning研究策略课程主要介绍宏基因组binning研究价值 、宏基因组个性化分析目标bin的挑选和研究思路,并结合水体微生物和抗药基因的案例进行解析;