基于NGS进行全基因组关联分析是当前QTL定位的最流行方法之一,关于动植物关联分析的系统课程较少,OS平台仅有一些简单的介绍。本课程将从原理、项目设计、分析方法和结果解读等方面全面讲授全基因组关联分析相关的内容,带领学员学习关联分析。
本程以实操为主(课程总时长12小时,实操时长8个小时,理论4小时)。并提供了课程系列相关的实操数据以及GWAS相关的附件下载。

课程内容:
课时1: 全基因组关联分析基本概念和材料选择
课程简介:主要介绍1)全基因组关联分析相关的基本概念;2)使用于关联分析的群体材料的选择,包括材料类型(自然群体材料、人工群体材料)、群体大小等。
课时2:表型考察处理与标记分型
课程简介:主要介绍1)不同类型表型的考察记录方法;2)表型的基本处理(正态性检验、异常值去除、多年多点表型的处理原则、表型数据的标准化、分类变量的哑变量赋值等);3)表型的基本分析(描述性统计分析、相关性分析、遗传力估计、BLUP);4)不同标记开发与分型方法简介与比较;5)基因型填补简介;6)基因型过滤的主要内容。
课时3: 群体结构和亲缘关系分析
课程简介:主要介绍:1)群体结构的评估方法:系统发育树、STRUCTURE、PCA;2)亲缘关系的评估方法:基于系谱关系推断亲缘关系、基于标记基因型推断亲缘关系
课时4: 关联分析模型和软件介绍
课程简介:主要介绍:1)常见的关联分析方法:卡方检验、GLM、MLM、MLMM等;2)常用的关联分析软件的简要介绍。

课时5: 结果解读与典型案例介绍
课程简介:主要介绍:1)关联分析的主要结果、正常情况、异常情况及异常问题的常见处理方法;2)结合GWAS的高分文章解析
课时6: 表型处理与标记筛选实操
课程简介:主要实操内容:1)表型的异常值去除、描述性统计图表的获得、BLUP值的求取、min-max和z-score标准化;2)基于VCF的基因型筛选实操,包括按质量值进行标记过滤、按分型完整度对标记进行过滤、按第二等位基因频率进行标记过滤、按哈迪温伯格平衡进行标记过滤等。
课时7:群体结构分析实操
课程简介:主要实操内容:1)基于VCF格式的基因型格式转换(转换为STRUCTURE、ADMIXTURE、smartpca格式);2)STRUCTURE(windows、linux)、ADMIXTURE(linux)的使用及结果解释与处理。
课时8:亲缘关系与PCA分析实操
课程简介:主要实操内容:1)使用gcta、ldak、tassel计算样本间的亲缘关系和进行PCA分析;2)使用spagedi计算样本间的亲缘关系; 3)使用smartpca进行PCA分析;4)亲缘关系结果的整理与展示。
课时9:关联分析实操
课程简介:主要实操内容:1)关联分析相关文件准备;2)使用plink进行case-control分析和逻辑回归(linux);3)使用tassel进行GLM/MLM/CMLM分析(windows、linux);4)其他软件如GAPIT、EMMAX、FaST-LMM部分命令行解释(linux)
课时10:结果筛选与展示
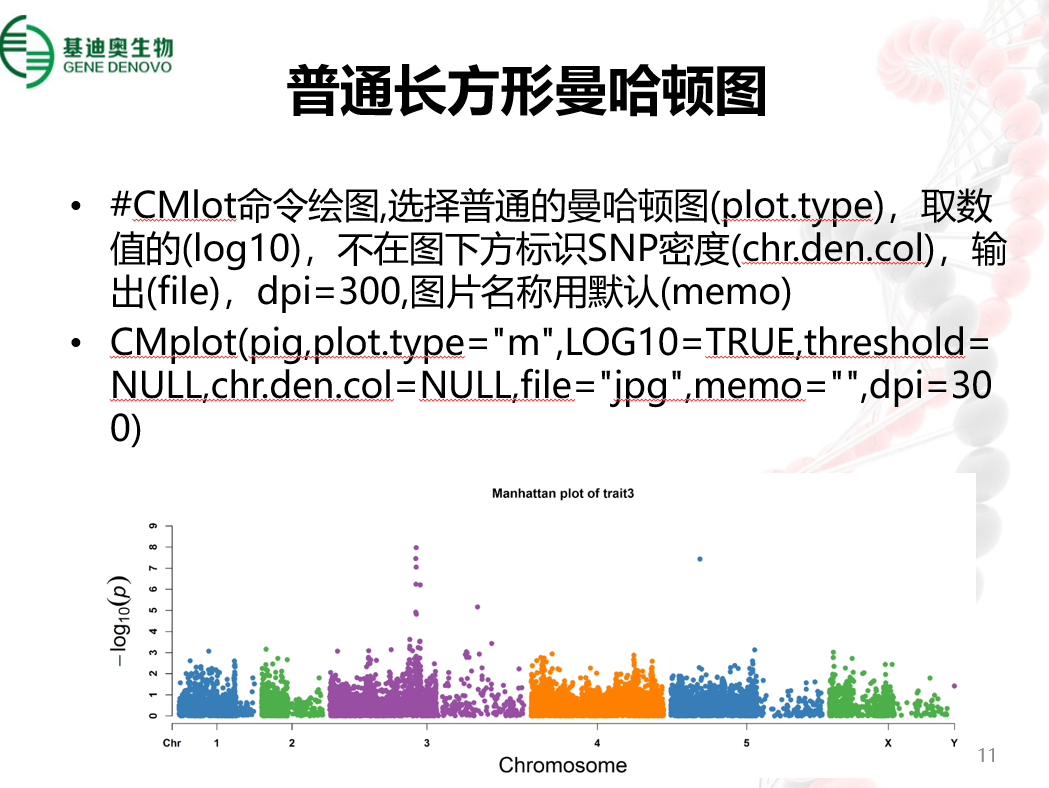
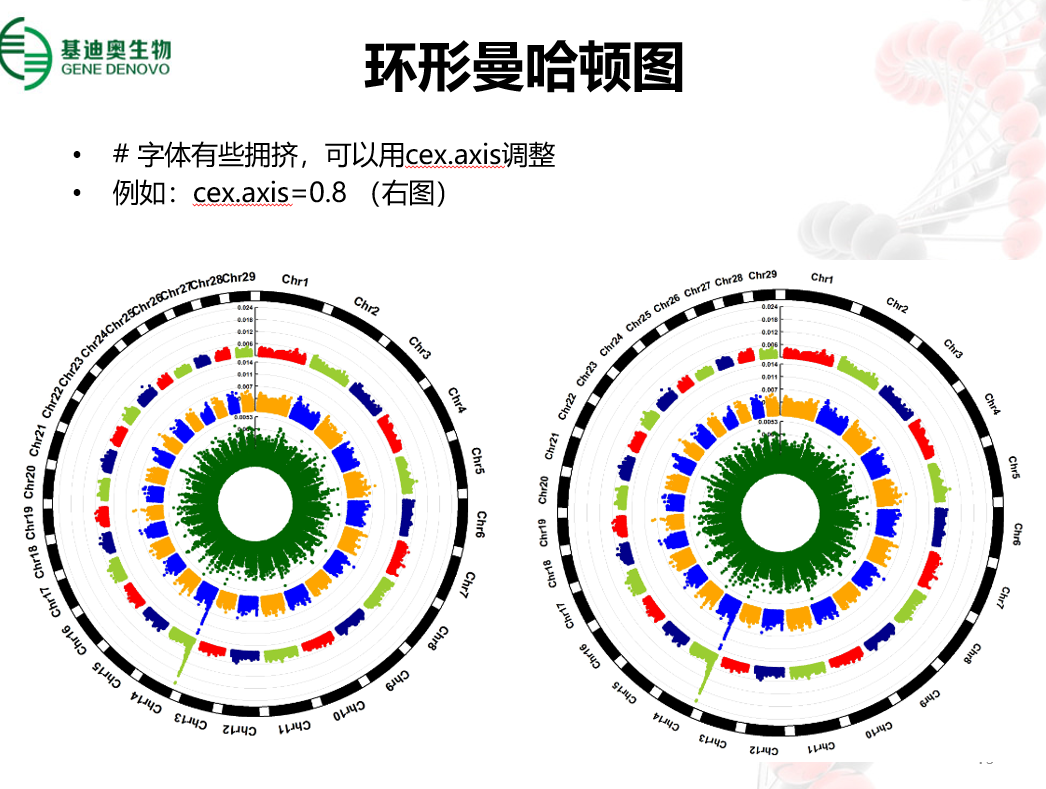
课程简介:主要实操内容:1)FDR调整;2)Bonferroni阈值的确定; 3)按给定阈值筛选显著位点;4)manhattan plot和quantile-quantile plot的绘制;5)λ的计算与结果解释。
课时11:LD衰减与LDblock分析
课程简介:主要实操内容:1)plink、Haploview计算LD;2)PopLDdecay计算LD并绘制衰减距离图;3)Haploview和LDheatmap计算并绘制LDblock。
温馨提醒:
1)OmicShare课程可以整系列购买,或单独购买子课程,具体课程页面查阅。
2)目前OmicShare的课程提供支付宝或微信付款;如果需要通过单位转账,请联系客服安排。
3)购买任意课程都可开发票,并且只开电子发票。有且仅开以下4种:技术咨询费、技术服务费、测序服务费。
4)购买视频课程,观看有效期为2年。
温馨提示:如有问题,请先在相应的小节视频页面里提问。

 扫码支付更轻松
扫码支付更轻松


















 广州大学城智慧谷A5座
广州大学城智慧谷A5座
 020-39341079
020-39341079
 omicshare@genedenovo.com
omicshare@genedenovo.com


